- [Review]
- Microbial and Enzymes That Can Regulate Plastic Degradation
Hwicheol Shin*
 , Sojin Eem*, Soyeon An*, Dongyeop X. Oh**,†
, Sojin Eem*, Soyeon An*, Dongyeop X. Oh**,†  , and Dong-Ku Kang*, ***, ****,†
, and Dong-Ku Kang*, ***, ****,† 
*Department of Chemistry, Incheon National University, Incheon 22012, Korea
**Department of Polymer Science and Engineering and Program in Environmental and Polymer Engineering, Inha University, Incheon 22212, Korea
***Bioplastic Research Center, Incheon National University, Incheon 22012, Korea
****Research Institute of Basic Sciences, Incheon National University, Incheon 22012, Korea- 생분해성 플라스틱 분해 조절을 위한 미생물 및 효소 기술
*인천대학교 화학과, **인하대학교 고분자공학과 및 환경 고분자공학 전공, ***인천대학교 바이오 플라스틱 센터, ****인천대학교 기초과학연구소
Reproduction, stored in a retrieval system, or transmitted in any form of any part of this publication is permitted only by written permission from the Polymer Society of Korea.
Biodegradable plastics have been developed as a replacement for non-degradable plastics to respond to climate change and achieve carbon neutrality and are growing in technological and industrial terms. However, the biodegradability of biodegradable plastics does not meet what is required from user aspects, and various demands are being made to control the decomposition speed and initiating time points. Therefore, multiple studies have recently been actively conducted to accelerate the decomposition of biodegradable plastics under aerobic and anaerobic conditions, particularly research on the discovery and improvement of biodegradable microorganisms and enzymes. In this paper, we aim to provide information for controlling the decomposition of biodegradable plastics by summarizing recent research on microorganisms and the enzymes.
생분해성 플라스틱은 기후변화 대응의 일환 그리고 탄소중립 실현을 위한 기존 난분해성 플라스틱 대체제로 개발되어 기술적, 산업적 측면에서 빠르게 성장하고 있다. 그러나, 산업적 퇴비화 조건과 달리 일반적인 환경은 생분해성 플라스틱이 충분한 분해속도를 가지기에 열악한 조건으로 기존 생분해성 플라스틱의 분해 속도 조절에 대한 다양한 요구가 이루어지고 있는 실정이다. 따라서, 최근 생분해성 플라스틱의 호기 및 혐기 조건에서의 분해 가속화를 위한 다양한 연구가 활발히 이루어지고 있으며, 특히 생분해 미생물 및 효소 발굴과 이의 개량에 대한 연구가 활발히 이루어지고 있다. 본 논문에서는 최근 이루어지고 있는 생분해성 플라스틱 분해 미생물 및 이의 효소에 대한 연구에 대한 정리를 통해, 생분해성 플라스틱 분해조절을 위한 정보를 제공하고자 한다.
Biodegradable plastics have been developed as a replacement for non-degradable plastics to respond to climate change and achieve carbon neutrality and are growing in technological and industrial terms. However, the biodegradability of biodegradable plastics does not meet what is required from user aspects, and various demands are being made to control the decomposition speed and initiating time points. We aim to provide information for controlling the decomposition of biodegradable plastics by summarizing recent research on microorganisms and the enzymes.
Keywords: biodegradable polymer, biodegradable plastic, microbial, enzyme, plastic digestion.
This research was financially supported by the Research Assistance Program (2020) of Incheon National University.
저자들은 이해상충이 없음을 선언합니다.
1907년에 최초로 합성된 플라스틱 고분자는 베이크라이트(Bakelite)로 세계 2차 대전 이후 군 외부에서 플라스틱이 널리 사용되면서 플라스틱 생산의 급속한 성장을 이루었다.1,2 플라스틱은 인공적으로 합성된 유기 폴리머로 정의되며, 주로 석유에서 유래한 비생분해성 물질로 낮은 열 및 전기전도도, 높은 내구성, 우수한 기계적 성질과 합리적인 가격을 제공한다.3 석유화학 플라스틱은 1930~1940년부터 상업적으로 이용되기 시작해 1950년대부터 대량 생산에 이르러, 지난 50년간 20배 증가하였다.4 2019년 연간 플라스틱 생산량은 3억 5900만 톤으로 전세계 누적 플라스틱 생산량은 2050년 말까지 260억 톤에 이를 것으로 예상된다.5 석유계 플라스틱의 뛰어난 기계적 물성, 가공성, 경량성은 소비재 제품의 성능을 단기간에 향상시켜주며 우리에게 사회적, 경제적 이익을 안겨주었다.6 현재 석유계 플라스틱은 패키징, 전자, 의료, 미용 등 다양한 산업에서 경량 및 유연성을 조절할 수 있는 범용성을 가진, 필수불가결한 소재이다.6
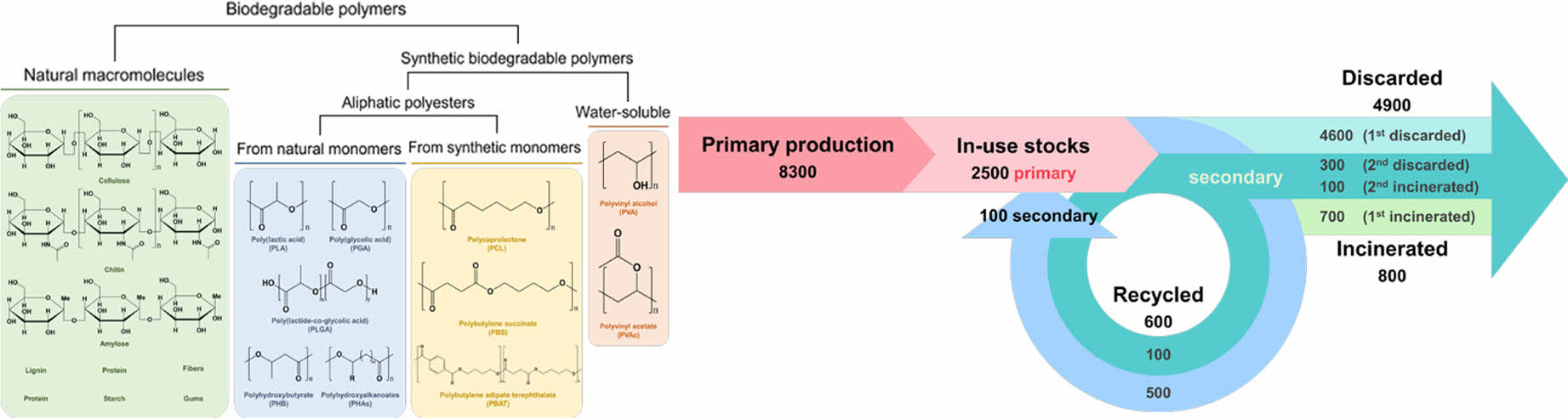
하지만, 혁명적이고 편리한 소재였던 플라스틱은 높은 안정성과 내화학성을 가지는 장점으로 인해 플라스틱 입자는 장기간 환경에 축적되어 점차 지구 환경을 위협하게 되었다.6 특히 해양에서는 현재 5조 개가 넘는 플라스틱 조각들이 다양한 크기와 모양으로 존재한다.7 따라서 여러 국가와 국제기구들이 플라스틱에 의한 환경 오염을 저감하기 위해 재활용을 권장하고 있음에도 불구하고, 전세계적으로 현재 플라스틱 재활용률은 10% 미만이며, 기존 폐기물 관리 인프라를 갖춘 선진국에서도 최대 30%에 머물러있다.2,8 1950년부터 2015년까지 생산된 플라스틱에 대한 매립, 소각, 재활용에 대한 자세한 수치 연구가 진행되었다. 2015년도까지 우리는 8300만톤의 플라스틱을 생산하였으며 사용 중인 것은 2500 만톤으로 추정된다. 이중 4900 만톤은 폐기되어 매립지 또는 자연 환경에 축적된다. 이외에 800 만톤(12%)은 소각되었고 600만톤(9%)가 재활용되었으며 단 10%만이 두 번 재활용되어 현재 플라스틱 폐기물의 재활용의 효과가 미미하다는 연구 결과를 얻었다(Figure 1).9 또한 매립된 플라스틱을 포함하여 완벽하게 분해되지 못한 플라스틱 폐기물은 매년 바다로 480만~1270만 톤이 유입되고 있으며, 미세 플라스틱(5 mm 미만)은 전체 해양 플라스틱 잔해 수의 90% 이상을 차지하고 있다.7,10 이들은 식품, 식수, 토양 및 공기 뿐만이 아니라 인간의 몸이나 주변의 퇴적물과 같이 예상치 못한 부분에서도 확인되고 있다.11 따라서 바이오 플라스틱으로도 알려진 생분해성 플라스틱은 석유화학 플라스틱이 환경과 인간에 미치는 해로운 영향을 완화하기 위한 솔루션으로 제안되었다.12
유럽 표준 EN 17228:2019에 따르면 바이오 플라스틱이란 바이오 기반이거나, 생분해성이거나 두 가지 특성을 모두 갖춘 플라스틱 소재이다.13 이 중 생분해성 플라스틱은 포장 및 일회용 품목과 농업용 덮개 필름에 많이 사용되며, 미생물 작용에 의해 이산화탄소, 메탄 및 미생물 바이오매스로 전환될 수 있는 플라스틱이다.14 플라스틱을 분해하는 미생물은 토양, 물, 공기, 퇴비 등 자연환경에 존재하며 생화학적 변형과 효소 활동을 통한 결합 절단을 통해 불용성 고분자를 가용성이고 구조적으로 단순한 고분자 화합물로 분해하고 전환할 수 있게 하는 중요한 역할을 한다.14 또한 생분해성 플라스틱은 새로운 end-of-life(EOL) 옵션의 하나로 재활용 경로를 제시한다는 점에서 이점이 있다.15 생물 재활용은 호기성/혐기성 소화를 통해 생분해성 물질을 각각 퇴비와 이산화탄소 및 바이오가스로 전환한다.15 특히 혐기성소화(anaerobic digestion, AD)에서 생분해성 부분이 무산소 조건에서 변환한 바이오가스는 열병합 발전 장치에서 전력 생산에 이용하거나 업그레이드 후 가스 그리드 및 천연가스 차량에 공급할 수 있다.16 2023년 전체 바이오 플라스틱 생산량은 약 218만 톤이며 이중 생분해성 플라스틱은 약 114만 톤으로 50% 이상을 차지하고 있다.17
또한 2019년 EU에서 일회용 플라스틱 사용을 금지하는 지침이 생기면서, 앞으로 플라스틱을 대체할 바이오 플라스틱의 사용량은 더욱 증가할 것이다.18 하지만 복합한 구조와 바이오매스 구성으로 인해 생분해성 플라스틱으로 분류되는 것도 쉽게 생분해되지 않는다.19 일례로 전 세계 바이오 플라스틱 생산량의 가장 큰 부분(25%)을 차지하는 poly(lactic acid)(PLA)는 퇴비화를 통해 15%만 분해되는 것으로 나타났으며 완전한 분해에 7.2년이 걸릴 것으로 추정된다.19 또한 실제로 생분해 속도는 바이오 플라스틱마다 다르며, 분해되는 외부 환경 요인, 블렌드 및 복합재의 특성에도 많은 관련이 있다.20 이들은 생분해성 플라스틱이 자연계에서 분해가 이루어짐에도 불구하고, 이들의 분해속도나 소비자나 환경적 측면에서 충분하지 못하다는 것을 말한다.
이에 미생물을 이용해 퇴비 내 퇴비화 가능한 플라스틱의 생분해를 가속화하는 연구가 많이 진행되었다. 미생물 활동과 퇴비화 과정은 각각의 온도, pH, 통기, 수분, 산소 유무와 크게 연관되며, 각 플라스틱에 적절한 미생물 군집과 효소들이 다르다.21 즉, 생분해의 향상에 핵심적인 미생물 및 효소 개발에 대한 연구가 이 분야의 핵심이라 할 수 있다. 이에 따라, 본 논문에서는 다양한 생분해성 고분자의 분해에 관여하는 미생물 및 효소에 대한 최근 연구동향을 정리하고자 한다.

|
Figure 1 Global production, use, and fate of polymer resins, synthetic fibers, and additives (1950 to 2015; in million metric tons).2 |
현재 많은 양의 플라스틱 폐기물이 환경에 버려지고 있고, 대부분이 미생물 분해에 저항하는 비생분해성 플라스틱으로 심각한 환경 문제가 나타나고 있다.22 최근 연구진들은 친환경적, 생분해성 및 무공해 소재를 생산하고자 노력하고 있으며, 우수한 상용성과 생분해성을 지닌 천연 및 합성 생분해성 플라스틱이 생산되고 있다.23,24
생분해성 플라스틱의 그 기원과 합성 방법, 화학적 조성, 가공 방법 및 용도에 따라 분류된다.24
생분해성 플라스틱은 천연 고분자와 합성 고분자로 분류가 되며, 합성 고분자는 다시 바이오매스(천연물) 기반과 석유계 기반으로 분류된다 (Figure 2). 천연 고분자 및 바이오매스 기반 플라스틱은 동물, 식물, 해양 및 미생물과 같은 생물학적 환경 요소에서 합성 및 추출하며, 석유계 기반 플라스틱은 구성 요소 중 일부가 석유에서 추출하여 제조한다.23,25,26
천연 고분자. 천연 고분자는 천연 고무, 리그닌 및 다당류 등과 같이 살아있는 생물에 의해 생산되거나 자연에서 직접 추출되는 고분자이다. 다당류에는 지구상에 가장 풍부한 천연 고분자로 알려진 셀룰로오스, 키틴, 아밀로오스 등이 포함되어 있으며, 각각 목화, 갑각류 껍질 및 감자 등에서 주로 얻을 수 있다.27 또한, 알긴산나트륨은 매우 중요한 다당류로서 하이드로겔 제조 및 의료분야에 널리 사용되고 있다.28,29
리그닌은 다당류 다음으로 풍부한 생체 고분자로, 침엽수나 활엽수 등의 목질부를 구성하는 지용성 페놀 고분자이다. 최근 많은 연구에서는 생분해성 열가소성 수지의 부족한 물성을 개선하기 위해, 내열성이 높은 리그닌을 화학적으로 결합하거나 복합화하여 물성을 향상시키는 개발에 집중하고 있다.30
이와 같이 연구가 진행됨에 따라 여러 천연 고분자들의 개발이 크게 증가될 것이며, 이를 기반으로 여러 제품들이 개발되어 우리 실생활에 크게 도움이 될 것으로 예상된다.
지방족 폴리에스터(Aliphatic Polyester). 폴리에스터는 구조적 특성으로 단단함, 유연함, 소수성 및 친수성 등 여러 성질을 보유하고 있어 매우 중요한 소재이다. 또한, 폴리에스터는 가수분해(hydrolysis; 에스터 결합의 가수분해 및 생물학적 촉매작용)31,32 또는 알코올분해(alcholysis; 용매분해)로 쉽게 올리고머(Oligomer) 및 단량체(Monomer)로 해중합이 가능하여, 재활용 기술 개발을 선도하는 소재이다. 이러한 장점으로 분해성 포장재로 사용되며 의학, 약리학 및 농업 등 특정 분야에서 일회용 제품으로 자주 사용되고 있다.33,34 지방족 폴리에스터는 원료의 생산 단계에서 크게 바이오매스 또는 석유계 기반로 분류된다.35,36
바이오매스 기반 생분해성 플라스틱. 바이오매스 기반 단량체로부터 합성된 지방족 폴리에스터로는 대표적으로 PLA, poly(glycolic acid)(PGA), Poly(lactide-co-glycolic acid)(PLGA), polyhydroxybutyrate(PHB) 및 Polyhydroxyalkanoates(PHAs) 등이 있다.
감자 전분, 밀, 쌀겨, 옥수수 및 바이오매스와 같은 재생 가능한 원료로부터 합성되는 PLA는 가장 대표적인 바이오매스 기반 생분해성 플라스틱으로, 생분해성 및 생체적합성이 높은 것으로 알려져 있다.37-39 PLA는 열가소성 선형 지방족 폴리에스터인 젖산(lacitc acid, LA)을 단량체로 합성되는데, 젖산은 L형(L-LA)과 D형(D-LA)을 거울상 이성질체로 가진다.40,41 고분자 사슬의 L-LA와 D-LA의 비율과 분포에 따라 PLA의 열적, 기계적 특성이 크게 달라진다.42 기본적으로 PLA는 L-LA가 주를 이루고 있어 반결정성 고분자인 poly(L-lactic acid)(PLLA)를 생성하며, D-LA의 비율이 높아지면 비정질 고분자인 poly(DL-lactic acid)(PDLLA)를 생성한다.43 이와 같은 특성으로 PLA는 다양한 응용 분야에서 사용되고 있다.44
PGA는 지방족 폴리에스터 중 가장 단순한 선형 구조를 가지고 있는 바이오매스 기반 생분해성 플라스틱이다.35 PGA는 PLA와 구조가 유사하지만 열변형 온도가 더 높고, 기계적 특성이 우수하며 특히 가스 차단성이 매우 높다.45 글리콜산 단량체(glycolic acid, GA)는 L-LA 및 D-LA와 공중합되어 PLGA를 생성하며, PLA와 PGA의 결합 중합에서 단량체 비율(L/G 비율)에 따라 생분해 속도에 영향으로 주며, L/G 비율이 증가할수록 분해 속도가 감소한다.39,46
PHAs는 다양한 미생물에 의해 합성되는 바이오매스 기반 생분해성 플라스틱이다. PHA는 1926년 Lemogine에 의해 처음 발견되었으며, 생분해성, 생체 적합성이 매우 뛰어난 열가소성 소재이며 다양하게 재생 가능 자원으로 개발할 수 있다. PHA는 난분해성 플라스틱인 polyethylene(PE)와 polypropylene (PP)의 잠재적인 대안으로 PHB는 이 계열의 가장 대표적인 플라스틱이다.23,47-49
석유계 기반 생분해성 플라스틱. 석유계 기반 단량체로부터 합성된 지방족 폴리에스터로는 대표적으로 polycaprolactone (PCL), polybutylene succinate(PBS) 및 poly(butylene adipate-co-terephthalate)(PBAT)등이 있다.
Caprolactone(CL)의 개환(ring-opening) 중합 반응으로 합성된 PCL은 선형 지방족 폴리에스터로, 석유계 기반 생분해성 플라스틱이다. PCL의 분해 속도는 결정화도 수준과 분자량에 의해 달라지며, 장점으로 호기성 이외에 혐기성 미생물의 활동으로도 분해된다.38 유리전이온도(glass transition temperature, Tg)가 -60 ℃로 매우 낮아 기계적 특성이 매우 우수한 생분해성 플라스틱이다. PCL은 치과 임플란트
용 cyclosporine을 포함한 나노입자 형태의 약물 전달체 제작 등에 사용되고 있다.50
PBS는 succinic acid(숙신산), diethyl succinate 또는 dimethyl succinate와 1,4-butanediol의 중축합 또는 개환 중합 반응에 의해 제조되는 석유계 기반 생분해성 플라스틱이다.51 녹는점(Tm)이 90-120 ℃인 열가소성 결정성 물질이며, 가수분해 메커니즘으로 천천히 분해 된다.23,38
PBS는 일반적으로 adipate와 같은 원료와 혼합되어 polybutylene succinate adipate(PBSA) 고분자를 형성하며, PBS보다 분해속도가 더 빠를 수 있다.23
PBAT는 butanediol(BDO), adipic acid(AA) 및 terephthalic acid의 중축합을 통해 합성되는 석유계 기반 생분해성 플라스틱이다. PBAT를 합성하려면 긴 반응시간, 높은 수준의 진공 및 190 ℃ 이상의 가열이 필요하다. 이러한 조건은 축합 반응을 촉진하고 수분을 제거하는데 기여한다.52,53 PBAT는 60 ℃ 퇴비에서 최대 약 PTA 함량의 50 mol%까지 분해되며, 생분해 속도는 고분자 내 PTA 함량에 따라 달라진다는 것이 밝혀졌다.54-56
수용성 합성 고분자(Water-Soluble Synthetic Polymers).Polyvinyl acetate(PVAc)는 vinyl acetate를 단량체로 자유 라디칼 중합 절차를 통해 합성된 수용성 합성 고분자이다.57 높은 유연성과 낮은 독성으로 식품 산업에서 사용되고 있으며, 특히 아시아 지경에서 과일과 채소를 코팅하는데 사용되고 있다.58 Poly(vinyl alcohol)(PVA)은 Polyvinyl acetate(PVAc)를 가수분해하여 제작되는 수용성 합성 고분자이며, 호기성 및 혐기성 조건에서 모두 생분해된다.59,60 PVA와 PVAc를 다른 석유계 생분해 플라스틱과 따로 분류하는 또 다른 이유는, 석유계 폴리에스터는 가수분해 가능한 에스터 그룹을 고분자 주쇄에 포함하고 있는 반면, PVA와 PVAc는 주쇄가 오직 C-C 결합으로만 이루어져 있기 때문이다.

|
Figure 2 Schematic shows biodegradable polymers classification based on their origin and method of production. |
플라스틱이 분해되는 단계는 크게 2단계로 구분된다. 첫번째 단계는 고분자 사슬이 극성 사슬 말단을 갖는 올리고머(oligomer)와 단량체(monomer)로 분해되는 단편화(fragmentation) 단계이며, 이 단계는 고분자 주사슬의 화학구조 및 분자 배치 등이 환경에 따라 가수 분해 또는 산화됨에 따라 발생한다. 두번째 단계는 곰팡이, 박테리아 등의 미생물에 의해 올리고머와 단량체가 분해되어, 호기성 또는 혐기성 환경에 따라 이산화탄소, 메탄가스, 물 및 바이오매스 등을 형성하는 것으로 이 단계를 생분해(biodegradation)라 한다(Figure 3).61,62
PLA. 환경에 따라 PLA가 분해되는 정도 및 속도가 달라지기 때문에 분해 조건을 어떻게 설정하는지가 중요하다. PLA는 pH, 온도, 습도, 산소 유무 등 다양한 요소에 의해 영향을 받는다. PLA의 분해는 생분해 능력을 가지는 미생물과 함께 배양하여 이루어진다(Table 1).63 일반적으로 온도가 증가함에 따라 PLA 생분해가 향상된다.64 Psuedonocardia sp. RM423는 방선균으로 PLA 생분해 시험 결과 토양에서 PLA 시트 분해가 중온성 온도보다 호열성 온도에서 더 빠르게 진행된다.65 이는 PLA의 가수분해와 관련이 있다. 미생물에 의해 분해가 이루어지기 전에 주로 화학적 가수분해로 인한 PLA와 같은 고분자의 분자량 감소 나타나는데 이는 온도에 따라 달라진다.66,67 고온에서 폴리머에 수분 흡착을 통한 가수분해의 진행을 돕는다.64
호열성 온도에서 Geobacillus thermocatenulatusstrain 41,68 Geobacillus thermoleovorans,69 Thermopolyspora flexuosaDSM 4318670 등이 있으며, 중온성 온도에서 Bacillus licheniformis,71 Pseudomonas aeruginosa strain S3&S4,72 Stenotrophomonas pavanii CH173 이 PLA를 분해하는 능력을 가지고 있다. 또한 Saccharothrix waywayandensis는 4일만에 100 mg PLLA 필름을 95 mg 이상 분해하였다.74
PLA를 분해하는 미생물이 활성을 보이는 효소는 protease, lipase, cutinase 등이 있다(Table 2). 그 중 PLA를 분해하는 미생물이 가장 많은 활성을 보이는 효소는 protease이다.75 PLA 분해 미생물은 효소활성에 의해 가수분해가 일어나 ester 결합을 절단하며, lactic acid 단량체를 생성한다.75 Protease는 n-butyl, ethyl-d, L-lactate를 가수분해할 수 있는것과 다르게, PDLA는 활성 부위에 수용될 수 없거나, acyl 효소 중간체를 만들 수 없기 때문에 PDLA는 가수분해가 불가능하다.76 반면, lipase는 PLLA보다 PDLA에서 활성 부위를 잘 인식하기 때문에 PDLA를 더 빠르게 분해할 수 있다.76
PHAs. PHA의 가장 중요한 특징은 높은 생분해성과 분해 후 부산물이 무독성이라는 것이다.115 PHA의 생분해성은 수분, 온도, pH, 미생물 집단, 표면적 및 결정화도 등 많은 요인의 영향을 받는다.116-118 또한 PHA는 호기성 및 혐기성 조건에서 분해가 가능하며, 염분 환경에서도 빠르게 분해되는데, 이는 해양 조건에서도 분해되는 것을 의미한다. 이를 통해 PHA는 환경에 해를 끼치지 않고 쉽게 폐기가 가능하여 중요한 생분해성 플라스틱이다.116
PHA를 분해하는 미생물은 매우 다양하며, 비교적 낮은 온도에서 생분해가 가능한 것을 확인하였다(Table 3). Alcanivorax sp.는 해양 균주로, PHA를 유일한 탄소원으로 사용하여 15 ℃와 22 ℃에서 60일 동안 생분해 시 분해 산물이 방출되었고, SEM을 통해 표면의 형태학적 변화를 확인했다.119 토양에서 분리된 Streptomyces sp.IN1은 30 ℃에서 P(3HB-co-3HV)를 분해하는 thermoalkanophilic P(3HB-co-3HV) esterase를 생산했다.120 혐기성 소화는 메탄 생성을 통해 가치화 된 에너지를 회수할 수 있다.121 하지만 생분해성 고분자의 혐기성 소화와 관련된 미생물에 관해서는 호기성 생분해에 비해 덜 연구되었다.122
Ilyobacter delafieldii는 호수의 혐기성 퇴적물에서 분리되었고, 최초로 분리된 혐기성 PHA 분해 균주이며 비교적 낮은 온도(20 ℃ 이하)에서 80% 이상의 분해를 보였다.123 이외에도, PHB 혐기성 분해 균주인 Enterobacter sp.와 메탄 생산이 관계 있는 것이 확인되었다.122 PHA의 효소적 생분해 메커니즘은 미생물에서 분비된 PHA depolymerase가 PHA를 수용성 올리고머와 단량체로 분해하는 것이며(Table 4), 분해 산물이 세포 내에서 에너지원으로 다시 대사된다.118
PGA. PGA는 건조한 공기나 진공 상태에서는 70 ℃에서 분해되지 않았으나, 70 ℃의 습한 조건에서 PGA 샘플의 분해는 70 ℃의 물에서 샘플의 분해와 유사하며 분해 정도가 높아지는 모습을 보인다.139 자연환경 중의 수증기(수분)가 PGA 분해의 주요 원인이며, 온도가 높을수록 분해 과정이 가속화되어 PGA의 유효기간 및 수명이 단축된다는 사실을 알 수 있다.139
PGA의 분해는 주로 호기성에서 이루어진다(Table 5). Flavobacterium polyglutamicum는 PGA 가수분해 효소(Table 6)를 이용하여 호기성, 28 ℃, pH 7조건에서 최적의 분해능력을 보였으며, Myrothecium sp.는 호기성, 37 ℃, pH 5조건에서 PGA를 분해하는 모습을 보였다.140,141 또 다른 미생물로는 B. licheniformis 9945a가 있을 수 있으며 호기성, 37 ℃에서 PGA를 가장 잘 분해하는 것으로 보고되어 있다.142
PLGA. PLGA를 분해하는 미생물은 아직 많은 연구가 진행되지 않은 것으로 보이며, 반응 조건 및 분석 시스템의 표준화가 부족하여 이들 연구에 사용된 효소의 분해조건을 비교하는 연구가 필요하다.142
PBS. PBS를 분해하는 미생물은 호기성 하에서만 발견이 되었는데(Table 7), Phyllophora antarctica에 의해 PBS필름은 토양실험에서 6주만에 60% 무게감소가 나타났다.143
Fusarium sp. FS1301는 21일만에 PBS의 필름 전체 중량에서 80%의 무게감소를 보였다.144
SEM 결과에서 많은 다공성을 가진 표면을 보였고, DSC 분석에 따라 PBS의 결정질과 비결정질 구조에 모두 손상을 입힌 것을 알 수 있었다.143 또한, Cryptococcus sp.에서 생산된 lipase에 의해 72시간과 16시간이라는 짧은 기간 내에 PBS와 PBSA가 완전히 분해된 것을 통해 lipase가 PBS 분해에 관여하는 효소라는 것을 확인할 수 있다(Table 8).144
PBAT. PBAT는 생분해성 멀칭 필름에 가장 많이 사용되어, PBAT를 분해하는 중온성 균을 발견하는 것이 현장 분해에 관련하여 중요하다. PBAT를 분해하는 호기성 균은 일반적으로 토양 환경에서 관찰된다(Table 9).
토양 환경에서 Bacillus pumilus와 밀접한 관련이 있는 3개의 PBAT 분해 균주인 NKCM3201, NKCM3202 및 NKCM3101을 분리하였다. 이 연구는 호기성 중온성 박테리아의 PBAT 가수분해 효소에 대한 첫번째 보고라는 것에 의의가 있다.151
농경지 토양에서 발굴한 Stenotrophomonas sp. YCJ1은 lipase 생산하여 PBAT 필름을 분해하는 것으로 나타났다(Table 9).149
이외에도 식물 성장에 영향을 미치지 않고 PBAT를 분해하는 Candida antarctica와 다양한 사슬 길이의 PBAT를 분해할 수 있는 Humicola insolens 및 Thermobifida cellulosilytica의 cutinases에 의해 가수분해가 일어나는 것으로 나타났다.152,153
혐기성 미생물인 Clostridium botulinum ATCC 3502(Cbotu_EstA 및 Cbotu_EstB)의 esterase는 PBAT를 가수분해하며, 혐기성 환경에서 효소에 의한 가수분해가 PBAT 생분해의 주요 메커니즘이라 설명한다(Table 10).153
PCL. PCL을 분해하는 균주는 호기 조건의 다양한 지역에서 발굴되었다(Table 11). PCL 분말 1 g이 함유된 배지는 대만 토양에서 발굴한 Streptomyces thermoviolaceus subsp.thermoviolaceus 76T-2에 의해 6시간만에 완전히 분해되었다.163
Pseudomonas pachastrellae JCM12285 균주는 PCL을 최대 1.39 ± 0.09 mg·cm−2·day−1 속도로 분해하였으며, 해수의 플라스틱 잔해에서 분리해낸 최초의 PCL 분해 균주이다. 이는 해양에서의 플라스틱 분해 가능성을 보여준다.164 혐기 슬러지에서 PCL 분해를 확인한 연구가 있었지만 생분해가 거의 일어나지 않는 결과를 통해 혐기 조건에서의 PCL 분해는 어려움을 밝혔다.165 PCL 분해에 관여하는 효소는 대부분 esterase의 일종으로 lipase와 cutinase 등(Table 12)이 있다.164,166
수용성 합성 고분자. PVAc는 주로 호기성 조건에서 생분해가 이루어지며, 토양에서 분리된 Penicillium 및 Aspergillus 등과 같은 곰팡이 종에 의해 분해된다(Table 13).175 PVA은 친수성, 낮은 독성, 기계적 성능 등으로 인해 최근 주목받고 있는 합성소재이다.176,179 Munzer Ullah 연구팀에 의하면, PVA는 이전까지 보고되지 않은 Stenotrophomonas sp. 균주에 의해 4일만에 90%까지 분해가 가능함을 밝혔다.177 또한 PVA 분해에 사용되는 효소는 질소원이 풍부할 경우 더 높은 분해효율을 보였다.177 Jianping Huang 연구팀은 호기조건이 아닌 혐기 조건에서 Betaproteobacteria 균주가 40 ℃에서 PVA 분해가 가능함을 입증하였다.178

|
Figure 3 Biodegradation steps of polymers (First step is fragmentation and the second step is the biodegradation by microbial). |
기존 난분해성 플라스틱의 높은 안정성 및 과도한 성장으로 플라스틱 폐기물은 장기간 환경에 축적되어 오염의 원인이 되었다. 이를 대체하고, 지속 가능한 순환 경제를 만들기 위해 생분해성 플라스틱에 대한 관심과 기술적, 산업적 요구가 빠르게 증가하고 있다. 그러나 생분해성 플라스틱은 상온에서 생분해도가 낮아 이의 폐기물의 분해에 다양한 공정이 요구되거나, 너무 높은 생분해도로 인해 실사용에서 요구하는 물성이 떨어지는 문제가 제기되고 있다. 이에 따라, 생분해 on/off와 같은 조절에 가능한 소재에 대한 요구가 이어지고 있으나, 관련 소재 개발은 요원한 실정이다. 본 논문에서는 다양한 생분해성 플라스틱에 대해 각각의 기계적 물성 및 열적 성질 등을 논의하고, 생분해성 플라스틱을 분해하는 호기 및 혐기성 미생물과 효소를 정리하여 생분해성 플라스틱 분해 조절에 대한 정보를 전달하고자 한다.
바이오매스 기반 생분해성 플라스틱인 PLA, PHA 및 PGA 등은 미생물이 주로 호기성 조건(토양, 퇴비 및 해양 등)에서 높은 생분해능을 가지며, 혐기성 조건(소화조, 하수 슬러지 및 혐기 퇴적 물 등)에서는 상대적으로 낮은 생분해도를 가지는 것으로 보고되고 있다. PLA의 경우 호열성 조건에서 높은 분해도를 가지며, 이의 분해에 protease류의 효소가 관여한다. PHA는 비교적 다양한 온도에서 분해되며, 특이하게도 Deltaproteobacteria 및 Ilyobacter delafieldii는 비교적 낮은 온도 및 혐기성 조건에서 PHA를 분해하는 것으로 보고되었다.
석유계 기반 생분해성 플라스틱인 PBS, PBAT 및 PCL 등은 대부분 호기성 조건에서 생분해능을 보이며, 혐기성 조건에서는 거의 분해되지 않는 것으로 보고되고 있다. 농사에 사용되는 멀칭 필름의 주 원료인 PBAT는 토양 환경에서 빠르게 분해되어야 하며, 이를 위해 호기성 중온성 미생물을 발굴하는 것이 중요하다. 하지만, 이의 분해에는 최소 수개월 이상 소요되며, 지역 토양 조건에 따라 분해에 소요되는 시간이 다양한 것으로 알려져 있다. 이에 따라, 사용자가 원하는 시점에서 분해 조절이 가능한 멀칭필름 소재 등에 대한 사용자로부터의 요구가 이루어지고 있는 실정이다.
본 논문에서 정리한 미생물 및 효소는 특정 조건(환경)에서 생분해성 플라스틱의 생분해도를 더욱 향상시키고, 이를 통한 생물학적 전처리 방법으로 활용 가능할 것으로 기대된다. 또한 이들을 활용한 생분해도 on/off를 포함하는 생분해 조절이 가능한 소재 개발에 적용 가능할 것으로 기대한다.
- 1. Shashoua, Y. Conservation of Plastics. Routledge: Danmark, 2008.
-

- 2. Geyer, R.; Jambeck, J. R. Production, Use, and Fate of All Plastics Ever Made. Sci. Adv. 2017, 3, e1700782.
-

- 3. Kibria, M. G.; Masuk, N. I. Plastic Waste: Challenges and Opportunities to Mitigate Pollution and Effective Management. Environ. Res. 2023, 17, 20.
-

- 4. Walker, T. R.; Fequet, L. Current trends of Unsustainable Plastic Production and Micro (nano) Plastic Pollution. Trac-Trends Anal. Chem. 2023, 160, 116984.
-

- 5. PlasticsEurope, The Circular Economy for Plastics—A European Overview. Technical Report 2019.
- 6. Padervand, M.; Lichtfouse, E. Removal of Microplastics from the Environment. A Review. Environ. Chem. Lett. 2020, 18, 807-828.
-

- 7. Eriksen, M.; Lebreton, L. C. Plastic Pollution in the World's Oceans: More Than 5 Trillion Plastic Pieces Weighing Over 250000 tons Afloat at Sea. PLoS One, 2014, 9, e111913.
-

- 8. Narancic, T.; Weirckx, N. Converting Petrochemical Plastic to Biodegradable Plastic, in The Handbook of Polyhydroxyalkanoates; CRC Press:Boca Raton, 2020.
-

- 9. Geyer, R.; Jambeck, J. R. Production, Use, and Fate of All Plastics ever Made. Sci. Adv. 2017, 3, e1700782.
-

- 10. Jambeck, J. R.; Geyer, R. Plastic Waste Inputs from Land Into the Ocean. Science 2015, 347, 768-771.
-

- 11. Chae, Y.; An, Y. Current Research Trends on Plastic Pollution and Ecological Impacts on the Soil Ecosystem: A Review. Environ. Pollut. 2018, 240, 387-395.
-

- 12. Ali, S. S.; Abdelkarim, E. A. Bioplastic Production in Terms of Life Cycle Assessment: A State-of-the-art Review. Env. Sci. Ecotechnol. 2023, 15, 100254.
-

- 13. EN 17228:2019 - European Standards. accessed April 28, 2020.
- 14. Pathak, V. M.; Navneet, J. B. Review on the Current Status of Polymer Degradation: a Microbial Approach. Bioresour. Bioprocess. 2017, 4, 15.
-

- 15. García-Depraect, O.; Bordel, S. Inspired by Nature: Microbial Production, Degradation and Valorization of Biodegradable Bioplastics for Life-cycle-engineered Products. Biotechnol. Adv. 2021, 53, 107772.
-

- 16. Prakash, O.; Mostafa, A. Upflow Anaerobic Sludge Blanket Reactor Operation Under High Pressure for Energy-rich Biogas Production. Bioresour. Technol. 2023, 376, 128897.
-

- 17. Bioplastics, E. Bioplastics Market Development Update 2023,2023 [cited 2024 05.23]; Available from: https://www.european-bioplastics.org/market/ (accessed May 23, 2024).
- 18. 2019/904, D. E. Directive (EU) 2019/904 of the European Parliament and of the Council of 5 June 2019 on the Reduction of the Impact of Certain Plastic Products on the Environment 2019 [cited 2024 23 May]; Available from: https://eur-lex.europa.eu/eli/dir/2019/904/oj (accessed May 23, 2024).
-

- 19. Cucina, M.; De Nisi, P. Degradation of Bioplastics in Organic Waste by Mesophilic Anaerobic Digestion, Composting and Soil Incubation. Waste Manage. 2021, 134, 67-77.
-

- 20. Thakur, S.; Chaudhary, J. Sustainability of Bioplastics: Opportunities and Challenges. Curr. Opin. Green Sustain. Chem. 2018, 13, 68-75.
-

- 21. Ahsan, W. A.; Hussain, A. Biodegradation of Different Types of Bioplastics Through Composting—a Recent Trend in Green Recycling. Catalysts 2023, 13, 294.
-

- 22. Shivam, P. Recent Developments on Biodegradable Polymers and Their Future Trends. Int. J. Eng. Sci. 2016, 4, 17-26.
- 23. Ghanbarzadeh, B.; Almasi, H. Biodegradable Polymers. Biodegradation - Life of Science 2013, 141-185.
-

- 24. Jha, K.; Kataria, R. Potential Biodegradable Matrices and Fiber Treatment for Green Composites: A Review. AIMS Mater. Sci. 2019, 6, 119-138.
-

- 25. Ho, M.-P.; Wang, H. Critical Factors on Manufacturing Processes of Natural Fibre Composites. Compos. Part B: Eng. 2012, 43, 3549-3562.
-

- 26. Balaji, A. B.; Pakalapati, H. Natural and Synthetic Biocompatible and Biodegradable Polymers. Biodegradable and Biocompatible Polym. Compos. 2018, 286, 3-32.
-

- 27. Li, X.; Su, X. Multifunctional Smart Hydrogels: Potential in Tissue Engineering and Cancer Therapy. J. Mat. Chem. B 2018, 6, 4714-4730.
-

- 28. Sun, J.-Y.; Zhao, X. Highly Stretchable and Tough Hydrogels. Nature 2012, 489, 133-136.
-

- 29. Yu, Y.; Shang, L. Design of Capillary Microfluidics for Spinning Cell-laden Microfibers. Nat. Protoc. 2018, 13, 2557-2579.
-

- 30. Chen, Y. R.; Sarkanen, S. Biodegradable Lignin-Based Plastics. Biodegradable Polymers in the Circular Plastics Economy 2022, 329-368.
-

- 31. Sriyapai, P.; Chansiri, K. Isolation and Characterization of Polyester-based Plastics-degrading Bacteria from Compost Soils. Microbiology 2018, 87, 290-300.
-

- 32. Sashiwa, H.; Fukuda, R. Microbial Degradation Behavior in Seawater of Polyester Blends Containing Poly(3-hydroxybutyrate-co-3-hydroxyhexanoate) (PHBHHx). Mar. Drugs 2018, 16, 34.
-

- 33. Sun, T.; Li, G. Suitability of Mulching with Biodegradable Film to Moderate Soil Temperature and Moisture and to Increase Photosynthesis and Yield in Peanut. Agric. Water Manage. 2018, 208, 214-223.
-

- 34. Koller, M.; Mukherjee, A. Polyhydroxyalkanoates (PHAs)–Production, Properties, and Biodegradation. In Biodegradable Polymers in the Circular Plastics Economy; Dusselier, M., Lange, J.-P., Eds.; Wiley: New Jersey, 2022; pp. 145-204.
-

- 35. Zhang, L.; Zhong, J.; Ren, X. Natural fiber-based biocomposites. In Green Biocomposites: Manufacturing and Properties; Jawaid, M., Sapuan, S. M., Alothman, O. Y., Eds.; Springer: Cham, 2017; pp 31-70.
-

- 36. Mangaraj, S.; Yadav, A. Application of Biodegradable Polymers in Food Packaging Industry: A Comprehensive Review. J. Packag. Technol. Res. 2019, 3, 77-96.
-

- 37. Jiang, L.; Sabzi, M. Biodegradable and Biobased Polymers. Applied Plastics Engineering Handbook 2024, 133-165.
-

- 38. Saini, R. Biodegradable Polymers. Appl. Chem. 2017, 13, 179-196.
- 39. Shah, T. V.; Vasava, D. A Glimpse of Biodegradable Polymers and Their Biomedical Applications. e-polymers 2019, 19, 385-410.
-

- 40. Muller, J.; González-Martínez, C. Combination of Poly(lactic) Acid and Starch for Biodegradable Food Packaging. Materials 2017, 10, 952.
-

- 41. Jem, K. J.; Tan, B. The Development and Challenges of Poly(lactic acid) and Poly(glycolic acid). Adv. Ind. Eng. Polym. Res. 2020, 3, 60-70.
-

- 42. Pawar, P. A.; Purwar, A. H. Biodegradable Polymers in Food Packaging. Am. J. Eng. Res. 2013, 2, 151-164.
- 43. Pan, Y.; Farmahini-Farahani, M. An Overview of Bio-based Polymers for Packaging Materials. J. Bioresour. Bioprod. 2016, 1, 106-113.
- 44. Luckachan, G. E.; Pillai, C. K. S. Biodegradable Polymers-a Review on Recent Trends and Emerging Perspectives. J. Polym. Environ. 2011, 19, 637-676.
-

- 45. Jem, K.; Tan, B. The Development and Challenges of Poly(lactic acid) and Poly(glycolic acid). Adv. Ind. Eng. Polym. Res. 2020, 3, 60-70.
-

- 46. Gentile, P.; Chiono, V. An Overview of Poly(lactic-co-glycolic) Acid (PLGA)-based Biomaterials for Bone Tissue Engineering. Int. J. Mol. Sci. 2014, 15, 3640-3659.
-

- 47. Aamer Ali Shah, Fariha Hasan, Biological Degradation of Plastics: a Comprehensive Review. Biotechnol. Adv. 2008, 26, 246-265.
-

- 48. Bakhtiari, S. S. E.; Karbasi, S. Modified Poly(3-hydroxybutyrate)-based Scaffolds in Tissue Engineering Applications: A Review. Int. J. Biol. Macromol. 2021, 166, 986-998.
-

- 49. Tebaldi, M. L.; Maia, A. L. C. Poly(-3-hydroxybutyrate-co-3-hydroxyvalerate) (PHBV): Current Advances in Synthesis Methodologies, Antitumor Applications and Biocompatibility. J. Drug Deliv. Sci. Technol. 2019, 51, 115-126.
-

- 50. Mukherjee, C.; Varghese, D. Recent Advances in Biodegradable Polymers–Properties, Applications and Future Prospects. Eur. Polym. J. 2023, 112068.
-

- 51. Rafiqah, S.; Khalina, A. A Review on Properties and Application of Bio-based Poly(butylene succinate). Polymers 2021, 13,1436.
-

- 52. Sousa, A. F.; Vilela, C. Biobased Polyesters and Other Polymers from 2,5-furandicarboxylic Acid: a Tribute to Furan Excellency. Polym. Chem. 2015, 6, 5961-5983.
-

- 53. Díaz, A.; Katsarava, R. Synthesis, Properties and Applications of Biodegradable Polymers Derived from Diols and Dicarboxylic Acids: from Polyesters to Poly(ester amide)s. Int. J. Mol. Sci. 2014, 15, 7064-7123.
-

- 54. Witt, U.; Muller, R. Biodegradable Polyester Copolymers with Adaptable Application Properties Based on Mass Chemical-Products. Chem. Ing. Tech. 1995, 67, 904-907.
- 55. Witt, U.; Müller, R. Biodegradation of Polyester Copolymers Containing Aromatic Compounds. J. Macromol. Sci. Part A-Pure Appl. Chem. 1995, 32, 851-856.
-

- 56. Witt, U.; Müller, R.-J. Evaluation of the Biodegradability of Copolyesters Containing Aromatic Compounds by Investigations of Model Oligomers. J. Environ. Polym. Degrad. 1996, 4, 9-20.
-

- 57. Leonard, E. Part 2, Wiley-lnterscience, New York, NY, High Polymers. 1971.
- 58. Kolter, K.; Dashevsky, A. Polyvinyl Acetate-based Film Coatings. Int. J. Pharm. 2013, 457, 470-479.
-

- 59. Marušincová, H.; Husárová, L. Polyvinyl Alcohol Biodegradation Under Denitrifying Conditions. Int. Biodeterior. Biodegrad. 2013, 84, 21-28.
-

- 60. Li, M.; Zhang, D. X. Enhancement of PVA-degrading Enzyme Production by the Application of pH Control Strategy. J. Microbiol. Biotechnol. 2012, 22, 220-225.
-

- 61. Lucas, N.; Bienaime, C. Polymer Biodegradation: Mechanisms and Estimation Techniques–A Review. Chemosphere 2008, 73, 429-442.
-

- 62. Chandra, R. U. S. T. G. I.; Rustgi, R. Biodegradable Polymers. Prog. Polym. Sci. 1998, 23, 1273-1335.
-

- 63. Doble, M. Biodegradation of Polymers. Indian J. Biotechnol. 2005, 4, 186-193.
- 64. Itävaara, M.; Karjomaa, S. Biodegradation of Polylactide in Aerobic and Anaerobic Thermophilic Conditions. Chemosphere 2002, 46, 879-885.
-

- 65. Apinya, T.; Sombatsompop, N. Selection of a Pseudonocardia sp. RM423 that Accelerates the Biodegradation of Poly(lactic) Acid in Submerged Cultures and in Soil Microcosms. Int. Biodeterior. Biodegrad. 2015, 99, 23-30.
-

- 66. Strömberg, E.; Karlsson, S. The Effect of Biodegradation on Surface and Bulk Property Changes of Polypropylene, Recycled Polypropylene and Polylactide Biocomposites. Int. Biodeterior. Biodegrad. 2009, 63, 1045-1053.
-

- 67. Rudnik, E.; Briassoulis, D. Comparative Biodegradation in Soil Behaviour of Two Biodegradable Polymers Based on Renewable Resources. J. Polym. Environ. 2011, 19, 18-39.
-

- 68. Tomita, K.; Nakajima, T. Degradation of Poly(L-lactic acid) by a Newly Isolated Thermophile. Polym. Degrad. Stabil. 2004, 84, 433-438.
-

- 69. Castro-Aguirre, E.; Auras, R. Enhancing the Biodegradation Rate of Poly(lactic acid) Films and PLA Bio-nanocomposites in Simulated Composting Through Bioaugmentation. Polym. Degrad. Stabil. 2018, 154, 46-54.
-

- 70. Husárová, L.; Pekařová, S. Identification of Important Abiotic and Biotic Factors in the Biodegradation of Poly(l-lactic acid). Int. J. Biol. Macromol. 2014, 71, 155-162.
-

- 71. Arena, M.; Abbate, C. Degradation of Poly(lactic acid) and Nanocomposites by Bacillus Licheniformis. Environ. Sci. Pollut. Res. 2011, 18, 865-870.
-

- 72. Satti, S. M.; Shah, A. A. Isolation and Characterization of Bacteria Capable of Degrading Poly(lactic acid) at Ambient Temperature. Polym. Degrad. Stabil. 2017, 144, 392-400.
-

- 73. Teeraphatpornchai, T.; Nakajima-Kambe, T. Isolation and Characterization of a Bacterium That Degrades Various Polyester-based Biodegradable Plastics. Biotechnol. Lett. 2003, 25, 23-28.
-

- 74. Jarerat, A.; Tokiwa, Y. Poly(L-lactide) Degradation by Saccharothrix Waywayandensis. Biotechnol. Lett. 2003, 25, 401-404.
-

- 75. Zaaba, N. F.; Jaafar, M. A Review on Degradation Mechanisms of Polylactic Acid: Hydrolytic, Photodegradative, Microbial, and Enzymatic Degradation. Polym. Eng. Sci. 2020, 60, 2061-2075.
-

- 76. Kawai, F.; Nakadai, K. Different Enantioselectivity of Two Types of Poly(lactic acid) Depolymerases Toward Poly(L-lactic acid) and Poly(D-lactic acid). Polym. Degrad. Stabil. 2011, 96, 1342-1348.
-

- 77. Jarerat, A.; Pranamuda, H. Poly(L-lactide)-degrading Activity in Various Actinomycetes. Macromol. Biosci. 2002, 2, 420-428.
-

- 78. Penkhrue, W., Khanongnuch, C., Isolation and Screening of Biopolymer-degrading Microorganisms from Northern Thailand. World J. Microbiol. Biotechnol. 2015, 31, 1431-1442.
-

- 79. Nakamura, K.; Tomita, T. Purification and Characterization of an Extracellular Poly(L-lactic acid) Depolymerase from a Soil Isolate, Amycolatopsis sp. Strain K104-1. Appl. Environ. Microbiol. 2001, 67, 345-353.
-

- 80. Chomchoei, A.; Pathom-Aree, W. Amycolatopsis Thailandensis sp. Nov., a Poly(L-lactic acid)-degrading Actinomycete, Isolated from Soil. Int. J. Syst. Evol. Microbiol. 2011, 61, 839-843.
-

- 81. Janczak, K.; Hrynkiewicz, K. Use of Rhizosphere Microorganisms in the Biodegradation of PLA and PET Polymers in Compost Soil. Int. Biodeterior. Biodegrad. 2018, 130, 65-75.
-

- 82. Shin, G.; Park, S.-A. A Micro-spray-based High-throughput Screening System for Bioplastic-degrading Microorganisms. Green Chem. 2021, 23, 5429-5436.
-

- 83. Yu, J.; Kim, P. D. Comparison of Polylactic Acid Biodegradation Ability of Brevibacillus Brevis and Bacillus Amyloliquefaciens and Promotion of PLA Biodegradation by Soytone. Biodegradation 2022, 33, 477-487.
-

- 84. Tomita, K.; Kuroki, Y. Isolation of Thermophiles Degrading Poly (L-lactic acid). J. Biosci. Bioeng. 1999, 87, 752-755.
-

- 85. Kim, M. N.; Kim, W. G. Poly(L-lactide)-degrading Activity of a Newly Isolated Bacterium. J. Appl. Polym. Sci. 2008, 109, 234-239.
-

- 86. Bonifer, K. S.; Wen, X. Bacillus Pumilus B12 Degrades Polylactic Acid and Degradation is Affected by Changing Nutrient Conditions. Front. Microbiol. 2019, 10, 2548.
-

- 87. Wang, Y.; Hu, T. Biodegradation of Polylactic Acid by a Mesophilic Bacteria Bacillus Safensis. Chemosphere 2023, 318, 137991.
-

- 88. Tomita, K.; Tsuji, H. Degradation of Poly(D-lactic acid) by a Thermophile. Polym. Degrad. Stabil. 2003, 81, 167-171.
-

- 89. Kim, M. N.; Park, S. T. Degradation of Poly(L-lactide) by a Mesophilic Bacterium. J. Appl. Polym. Sci. 2010, 117, 67-74.
-

- 90. Wu, C.-S. Renewable Resource-based Composites of Recycled Natural Fibers and Maleated Polylactide Bioplastic: Characterization and Biodegradability. Polym. Degrad. Stabil. 2009, 94, 1076-1084.
-

- 91. Hanphakphoom, S.; Maneewong, N. Characterization of Poly(L-lactide)-degrading Enzyme Produced by Thermophilic Filamentous Bacteria Laceyella Sacchari LP175. J. Gen. Appl. Microbiol. 2014, 60, 13-22.
-

- 92. Lomthong, T.; Chotineeranat, S. Production and Characterization of Raw Starch Degrading Enzyme from a Newly Isolated Thermophilic Filamentous Bacterium, Laceyella Sacchari LP175. Starch - Stärke 2015, 67, 255-266.
-

- 93. Pathom-aree, W.; Butbunchu, N. Bioprocess Optimization Platform for Valorization of Poly(lactic)-based Bioplastic Waste Using PLA-degrading Actinobacteria, Saccharothrix sp. MY1 Cultured in Silk Wastewater as Low-cost Nutrient Source. Biomass Conv. Bioref. 2022, DOI:10.1007/s13399-022-03524-8.
-

- 94. Nair, N. R.; Sekhar, V. C.; Augmentation of a Microbial Consortium for Enhanced Polylactide (PLA) Degradation. Indian J. Microbiol. 2016, 56, 59-63.
-

- 95. Jeon, H. J.; Kim, M. N. Biodegradation of Poly(L-lactide)(PLA) Exposed to UV Irradiation by a Mesophilic Bacterium. J. Appl. Polym. Sci. 2013, 85, 289-293.
-

- 96. Wang, Z.; Wang, Y. Purification and Characterization of Poly(L-lactic acid) Depolymerase from Pseudomonas sp. Strain DS04-T. Polym. Eng. Sci. 2011, 51, 454-459.
-

- 97. Yagi, H.; Ninomiya, F. Mesophilic Anaerobic Biodegradation Test and Analysis of Eubacteria and Archaea Involved in Anaerobic Biodegradation of Four Specified Biodegradable Polyesters. Polym. Degrad. Stabil. 2014, 110, 278-283.
-

- 98. Karamanlioglu, M.; Houlden, A. Isolation and Characterisation of Fungal Communities Associated with Degradation and Growth on the Surface of Poly(lactic) Acid (PLA) in Soil and Compost. Int. Biodeterior. Biodegrad. 2014, 95, 301-310.
-

- 99. Torres, A.; Li, S. Screening of Microorganisms for Biodegradation of Poly(lactic-acid) and Lactic Acid-containing Polymers. Appl. Environ. Microbiol. 1996, 62, 2393-2397.
-

- 100. Harmaen, A. S.; Khalina, A. Thermal and Biodegradation Properties of Poly(lactic acid)/fertilizer/oil Palm Fibers Blends Biocomposites. Polym. Compos. 2015, 36, 576-583.
-

- 101. Lipsa, R.; Tudorachi, N. Biodegradation of Poly(lactic acid) and Some of Its Based Systems with Trichoderma Viride. Int. J. Biol. Macromol. 2016, 88, 515-526.
-

- 102. Jarerat, A.; Tokiwa, Y. Degradation of Poly(L-lactide) by a Fungus. Macromol. Biosci. 2001, 1, 136-140.
-

- 103. Bubpachat, T.; Sombatsompop, N. Isolation and Role of Polylactic Acid-degrading Bacteria on Degrading Enzymes Productions and PLA Biodegradability at Mesophilic Conditions. Polym. Degrad. Stabil. 2018, 152, 75-85.
-

- 104. Sakai, K.; Kawano, H. Isolation of a Thermophilic Poly-L-Lactide Degrading Bacterium from Compost and Its Enzymatic Characterization. J. Biosci. Bioeng. 2001, 92, 298-300.
-

- 105. Hajighasemi, M.; Nocek, B. P. Biochemical and Structural Insights Into Enzymatic Depolymerization of Polylactic Acid and Other Polyesters by Microbial Carboxylesterases. Biomacromolecules 2016, 17, 2027-2039.
-

- 106. Liang, T.-W.; Jen, S.-N. Application of Chitinous Materials in Production and Purification of a Poly(L-lactic acid) Depolymerase from Pseudomonas Tamsuii TKU015. Polyemrs 2016, 8, 98.
-

- 107. Hoshino, A.; Isono, Y. Degradation of Aliphatic Polyester Films by Commercially Available Lipases with Special Reference to Rapid and Complete Degradation of Poly(L-lactide) Film by Lipase PL Derived From Alcaligenes sp. Biodegradation 2002, 13, 141-147.
-

- 108. Akutsu-Shigeno, Y.; Teeraphatpornchai, T. Cloning and Sequencing of a Poly(DL-lactic acid) Depolymerase Gene from Paenibacillus Amylolyticus Strain TB-13 and Its Functional Expression in Escherichia coli. Appl. Environ. Microbiol. 2003, 69, 2498-2504.
-

- 109. Pranamuda, H.; Tokiwa, Y. Polylactide Degradation by An Amycolatopsis sp. Appl. Environ. Microbiol. 1997, 63, 1637-1640.
-

- 110. Ikura, Y.; Kudo, T. Isolation of a Microorganism Capable of Degrading Poly(L-lactide). J. Gen. Appl. Microbiol. 1999, 45, 247-251.
-

- 111. Pranamuda, H.; Tokiwa, Y. Degradation of Poly(L-lactide) by Strains Belonging to Genus Amycolatopsis. Appl. Environ. Microbiol. 1999, 21, 901-905.
-

- 112. Pranamuda, H.; Tsuchii, A. Poly(L-lactide)-Degrading Enzyme Produced by Amycolatopsis sp. Macromol. Biosci. 2001, 1, 25-29.
-

- 113. Jarerat, A.; Tokiwa, Y. Production of Poly(L-lactide)-degrading Enzyme by Amycolatopsis Orientalis for Biological Recycling of Poly(L-lactide). Appl. Microbiol. Biotechnol. 2006, 72, 726-731.
-

- 114. Jarerat, A.; Tokiwa, Y. Poly(L-lactide) Degradation by Kibdelosporangium Aridum. Biotechnol. Lett. 2003, 25, 2035-2038.
-

- 115. Vert, M.; Doi, Y. Terminology for Biorelated Polymers and Applications (IUPAC Recommendations 2012). Pure Appl. Chem. 2012, 84, 377-410.
-

- 116. Muhammadi; Shabina; Afzal, M.; Hameed, S. Bacterial Polyhydroxyalkanoates-eco-friendly Next Generation Plastic: Production, Biocompatibility, Biodegradation, Physical Properties and applications. Green Chem. Lett. Rev. 2015, 8, 56-77.
-

- 117. Jendrossek, D.; Handrick, R. Microbial Degradation of Polyhydroxyalkanoates. Annu. Rev. Microbiol. 2002, 56, 403-432.
-

- 118. Numata, K.; Abe, H. Biodegradability of Poly(hydroxyalkanoate) Materials. Materials 2009, 2, 1104-1126.
-

- 119. Zhang, Y.; Cao, Y. Marine Biodegradation of Plastic Films by Alcanivorax Under Various Ambient Temperatures: Bacterial Enrichment, Morphology Alteration, and Release of Degradation Products. Sci. Total Environ. 2024, 917, 170527.
-

- 120. Zhou, W.; Bergsma, S. Polyhydroxyalkanoates (PHAs) Synthesis and Degradation by Microbes and Applications Towards a Circular Economy. J. Environ. Manage. 2023, 341, 118033.
-

- 121. Cho, J. Y., Park, S. L. Polyhydroxyalkanoates (PHAs) Degradation by the Newly Isolated Marine Bacillus sp. JY14. Chemosphere 2021, 283, 131172.
-

- 122. Cazaudehore, G.; Monlau, F. Active Microbial Communities During Biodegradation of Biodegradable Plastics by Mesophilic and Thermophilic Anaerobic Digestion. J. Hazard. Mater. 2023, 443, 130208.
-

- 123. Mas-Castella, J.; Urmeneta, J. Biodegradation of poly-β-hydroxyalkanoates in Anaerobic Sediments. Int. Biodeterior. Biodegrad. 1995, 35, 155-174.
-

- 124. Kita, K.; Mashiba, S.-I. Cloning of Poly(3-hydroxybutyrate) Depolymerase from a Marine Bacterium, Alcaligenes Faecalis AE122, and Characterization of Its Gene Product. Biochim. Biophys. Acta-Gene Struct. Expression 1997, 1352, 113-122.
-

- 125. Omura, T.; Isobe, N. Microbial Decomposition of Biodegradable Plastics on the Deep-sea Floor. Nat. Commun. 2024, 15, 568.
-

- 126. Kanmani, P.; Kumaresan, K. Enzymatic Degradation of Polyhydroxyalkanoate Using Lipase from Bacillus Subtilis. Int. J. Environ. Sci. Technol. 2016, 13, 1541-1552.
-

- 127. Volova, T.; Boyandin, A. Biodegradation of Polyhydroxyalkanoates (PHAs) in Tropical Coastal Waters and Identification of PHA-degrading Bacteria. Polym. Degrad. Stabil. 2010, 95, 2350-2359.
-

- 128. Phukon, P.; Saikia, J. P. Bio-plastic (P-3HB-co-3HV) from Bacillus Circulans (MTCC 8167) and Its Biodegradation. Colloid Surf. B-Biointerfaces 2012, 92, 30-34.
-

- 129. Focarete, M. L.; Ceccorulli, G. Further Evidence of Crystallinity-Induced Biodegradation of Synthetic Atactic Poly(3-hydroxybutyrate) by PHB-depolymerase A from Pseudomonas l Emoignei. Blends of Atactic Poly(3-hydroxybutyrate) with Crystalline Polyesters. Macromolecules 1998, 31, 8485-8492.
-

- 130. Mukai, K.; Yamada, K. Efficient Hydrolysis of Polyhydroxyalkanoates by Pseudomonas Stutzeri YM1414 Isolated from Lake Water. Polym. Degrad. Stabil. 1994, 43, 319-327.
-

- 131. Elbanna, K.; Lütke-Eversloh, T. Studies on the Biodegradability of Polythioester Copolymers and Homopolymers by Polyhydroxyalkanoate (PHA)-degrading Bacteria and PHA Depolymerases. Arch. Microbiol. 2004, 182, 212-225.
-

- 132. Mabrouk, M. M.; Sabry, S. A. Degradation of Poly(3-hydroxybutyrate) and Its Copolymer Poly(3-hydroxybutyrate-co-3-hydroxyvalerate) by a Marine Streptomyces sp. SNG9. Microbiol. Res. 2001, 156, 323-335.
-

- 133. Berezina, N.; Yada, B. Enzymatic Surface Treatment of Poly(3-hydroxybutyrate)(PHB), and Poly(3-hydroxybutyrate-co-3-hydroxyvalerate) (PHBV). J. Chem. Technol. Biotechnol. 2015, 90, 2036-2039.
-

- 134. Zadjelovic, V.; Chhun, A. Beyond Oil Degradation: Enzymatic Potential of Alcanivorax to Degrade Natural and Synthetic Polyesters. Environ. Microbiol. 2020, 22, 1356-1369.
-

- 135. Gricajeva, A.; Nadda, A. K. Insights Into Polyester Plastic Biodegradation by Carboxyl Ester Hydrolases. J. Chem. Technol. Biotechnol. 2022, 97, 359-380.
-

- 136. Allen, A. D.; Anderson, W. Isolation and Characterization of An Extracellular Thermoalkanophilic P(3HB-co-3HV) Depolymerase from Streptomyces sp. IN1. Int. Biodeterior. Biodegrad. 2011, 65, 777-785.
-

- 137. Abe, T.; Kobayashi, T. Properties of a Novel Intracellular Poly (3-hydroxybutyrate) Depolymerase with High Specific Activity (PhaZd) in Wautersia Eutropha H16. J. Bacteriol. 2005, 187, 6982-6990.
-

- 138. Leathers, T. D.; Govind, N. S. Biodegradation of Poly(3-hydroxybutyrate-co-3-hydroxyvalerate) by a Tropical Marine Bacterium, Pseudoalteromonas sp. NRRL B-30083. J. Polym. Environ. 2000, 8, 119-124.
-

- 139. Chen, S.; Zhang, X. Degradation of PGA, Prepared by Reactive Extrusion Polymerization, in Water, Humid, and Dry Air, and in a Vacuum. J. Mater. Res. 2020, 35, 1846-1856.
-

- 140. Volcani, B.; Margalith, P. A New Species (Flavobacterium polyglutamicum) Which Hydrolyzes the γ-L-glutamyl Bond in Polypeptides. J. Bacteriol. 1957, 74, 646-655.
-

- 141. Tanaka, T.; Hiruta, O. Purification and Characterization of Poly (γ-glutamic acid) Hydrolase from a Filamentous Fungus, Myrothecium sp. TM-4222. Biosci., Biotechnol., Biochem. 1993, 57, 2148-2153.
-

- 142. King, E. C.; Blacker, A. J. Enzymatic Breakdown of Poly-γ-D-Glutamic Acid in Bacillus Licheniformis: Identification of a Polyglutamyl γ-hydrolase Enzyme. Biomacromolecules 2000, 1, 75-83.
-

- 143. Kitamoto, H. K.; Shinozaki, Y. Phyllosphere Yeasts Rapidly Break Down Biodegradable Plastics. AMB Express 2011, 1, 1-11.
-

- 144. Mao, H.; Liu, H. Biodegradation of Poly(butylene succinate) by Fusarium sp. FS1301 and Purification and Characterization of Poly(butylene succinate) Depolymerase. Polym. Degrad. Stabil. 2015, 114, 1-7.
-

- 145. Maeda, H.; Yamagata, Y. Purification and Characterization of a Biodegradable Plastic-degrading Enzyme from Aspergillus Oryzae. Appl. Microbiol. Biotechnol. 2005, 67, 778-788.
-

- 146. Lee, S.-H.; Kim, M.-N. Isolation of Bacteria Degrading Poly (butylene succinate-co-butylene adipate) and Their Lip A Gene. Int. Biodeterior. Biodegrad. 2010, 64, 184-190.
-

- 147. Thirunavukarasu, K.; Purushothaman, S. Degradation of Poly (butylene succinate) and Poly(butylene succinate-co-butylene adipate) by a Lipase from Yeast Cryptococcus sp. Grown on Agro-industrial Residues. Int. Biodeterior. Biodegrad. 2016, 110, 99-107.
-

- 148. Hu, X.; Gao, Z. Enzymatic Degradation of Poly(butylene succinate) by Cutinase Cloned from Fusarium Solani. Polym. Degrad. Stabil. 2016, 134, 211-219.
-

- 149. Jia, H.; Zhang, M. Degradation of Poly(butylene adipate-co-terephthalate) by Stenotrophomonas sp. YCJ1 Isolated from Farmland Soil. J. Environ. Sci. 2021, 103, 50-58.
-

- 150. Gamerith, C.; Vastano, M. Enzymatic Degradation of Aromatic and Aliphatic Polyesters by P. pastoris Expressed Cutinase 1 from Thermobifida Cellulosilytica. Front. Microbiol. 2017, 8, 938.
-

- 151. Muroi, F.; Tachibana, Y. Characterization of a Poly(butylene adipate-co-terephthalate) Hydrolase from the Aerobic Mesophilic Bacterium Bacillus Pumilus. Polym. Degrad. Stabil. 2017, 137, 11-22.
-

- 152. Kanwal, A.; Zhang, M. Enzymatic Degradation of Poly (butylene adipate co-terephthalate) (PBAT) Copolymer Using Lipase B from Candida Antarctica (CALB) and Effect of PBAT on Plant Growth. Polym. Bull. 2022, 79, 9059-9073.
-

- 153. Perz, V.; Bleymaier, K. Substrate Specificities of Cutinases on Aliphatic–aromatic Polyesters and on Their Model Substrates. New Biotech. 2016, 33, 295-304.
-

- 154. Wang, A.; De Silva, K. Cr-Free Anticorrosive Primers for Marine Propeller Applications. Polymers 2024, 16, 408.
-

- 155. Perz, V.; Baumschlager, A. Hydrolysis of Synthetic Polyesters by Clostridium Botulinum Esterases. Biotechnol. Bioeng. 2016, 113, 1024-1034.
-

- 156. Tesei, D.; Quartinello, F. Shotgun Proteomics Reveals Putative Polyesterases in the Secretome of the Rock-inhabiting Fungus Knufia Chersonesos. Sci Rep 2020, 10, 9770.
-

- 157. Biundo, A.; Hromic, A. Characterization of a Poly(butylene adipate-co-terephthalate)-hydrolyzing Lipase from Pelosinus Fermentans. Appl. Microbiol. Biotechnol. 2016, 100, 1753-1764.
-

- 158. Wallace, P. W.; Haernvall, K. PpEst is a novel PBAT Degrading Polyesterase Identified by Proteomic Screening of Pseudomonas Pseudoalcaligenes. Appl. Microbiol. Biotechnol. 2017, 101, 2291-2303.
-

- 159. Yang, Y.; Min, J. Complete Bio-degradation of Poly(butylene adipate-co-terephthalate) via Engineered Cutinases. Nat. Commun. 2023, 14, 1645.
-

- 160. Xu, J.; Feng, K. Enhanced Biodegradation Rate of Poly (butylene adipate-co-terephthalate) Composites Using Reed Fiber. Polymers 2024, 16, 411.
-

- 161. Jamnongkan, T.; Sirichaicharoenkol, K. Innovative Electrospun Nanofiber Mats Based on Polylactic Acid Composited with Silver Nanoparticles for Medical Applications. Polymers 2024, 16, 409.
-

- 162. Kalia, V. C.; Patel, S. K. Manipulating Microbial Cell Morphology for the Sustainable Production of Biopolymers. Polymers 2024, 16, 410.
-

- 163. Chua, T.-K.; Tseng, M. Degradation of Poly(ε-caprolactone) by Thermophilic Streptomyces Thermoviolaceus Subsp. Thermoviolaceus 76T-2. Amb Express 2013, 3, 8.
-

- 164. Suzuki, M.; Tachibana, Y. Microbial Degradation of Poly(ε-caprolactone) in a Coastal Environment. Polym. Degrad. Stabil. 2018, 149, 1-8.
-

- 165. Federle, T. W.; Barlaz, M. A. Anaerobic Biodegradation of Aliphatic Polyesters: Poly(3-hydroxybutyrate-co-3-hydroxyoctanoate) and Poly(ε-caprolactone). Biomacromolecules 2002, 3, 813-822.
-

- 166. Sivalingam, G.; Vijayalakshmi, S. Enzymatic and Thermal Degradation of Poly(ε-caprolactone), Poly(D,L-lactide), and Their Blends. Ind. Eng. Chem. Res. 2004, 43, 7702-7709.
-

- 167. Oda, Y.; Oida, N. Polycaprolactone Depolymerase Produced by the Bacterium Alcaligenes Faecalis. FEMS Microbiol. Lett. 1997, 152, 339-343.
-

- 168. Budkum, J.; Thammasittirong, S. N.-R. High Poly ε-caprolactone Biodegradation Activity by a New Acinetobacter Seifertii Isolate. Folia Microbiol. 2022, 67, 659-669.
-

- 169. Hoang, K.-C.; Tseng, M. Degradation of Polyethylene Succinate (PES) by a New Thermophilic Microbispora Strain. Biodegradation 2007, 18, 333-342.
-

- 170. Nakasaki, K.; Matsuura, H. Synergy of Two Thermophiles Enables Decomposition of Poly-ɛ-caprolactone Under Composting Conditions. FEMS Microbiol. Ecol. 2006, 58, 373-383.
-

- 171. Al Hosni, A. S.; Pittman, J. K. Microbial Degradation of Four Biodegradable Polymers in Soil and Compost Demonstrating Polycaprolactone as an Ideal Compostable Plastic. Waste Manage. 2019, 97, 105-114.
-

- 172. Fields, R.; Rodriguez, F. Microbial Degradation of Polyesters: Polycaprolactone Degraded by P. pullulans. J. Appl. Polym. Sci. 1974, 18, 3571-3579.
-

- 173. Khatiwala, V. K.; Shekhar, N. Biodegradation of Poly(ε-caprolactone) (PCL) Film by Alcaligenes Faecalis. J. Polym. Environ. 2008, 16, 61-67.
-

- 174. Sivalingam, G.; Vijayalakshmi, S. Enzymatic and Thermal Degradation of Poly(ε-caprolactone), Poly(D,L-lactide), and Their Blends. Ind. Eng. Chem. Res. 2004, 43, 7702-7709.
-

- 175. Trejo, A. G. J. E.; Safety, E. Fungal Degradation of Polyvinyl Acetate. Ecotox. Environ. Safe. 1988, 16, 25-35.
-

- 176. Suleiman, G. S. A.; Zeng, X. Recent Advances and Challenges in Thermal Stability of PVA-based Film: A Review. Polym. Adv. Technol. 2024, 35, e6327.
-

- 177. Ullah, M.; Weng, C.-H. Degradation of Polyvinyl Alcohol by a Novel Bacterial Strain Stenotrophomonas sp. SA21. Environ. Technol. 2018, 39, 2056-2061.
-

- 178. Huang, J.; Yang, S. Performance and Diversity of Polyvinyl Alcohol-degrading Bacteria Under Aerobic and Anaerobic Conditions. Biotechnol. Lett. 2016, 38, 1875-1880.
-

- 179. Lee, J.-A.; Kim, M.-N. J. P. D. Isolation of New and Potent Poly (vinyl alcohol)-degrading Strains and Their Degradation Activity. Polym. Degrad. Stabil. 2003, 81, 303-308.
-

- 180. Matsumura, S.; Shimura, Y. Effects of Molecular Weight and Stereoregularity on Biodegradation of Poly(vinyl alcohol) by Alcaligenes Faecalis. Biotechnol. Lett. 1994, 16, 1205-1210.
-

- 181. Watanabe, Y.; Hamada, N. Purification and Properties of a Polyvinyl Alcohol-degrading Enzyme Produced by a Strain of Pseudomonas. Arch. Biochem. Biophys. 1976, 174, 575-581.
-

- 182. Ronkvist, Å. M.; Lu, W. Cutinase-catalyzed Deacetylation of Poly(vinyl acetate). Macromolecules 2009, 42, 6086-6097.
-

- 183. Fukae, R.; Fujii, T. Biodegradation of Poly(vinyl alcohol) with High Isotacticity. Polym. J. 1994, 26, 1381-1386.
-

- 184. Kim, B.; Sohn, C. Degradation of Polyvinyl Alcohol by Sphingomonas sp. SA3 and Its Symbiote. J. Ind. Microbiol. Biotechnol. 2003, 30, 70-74.
-

- 185. Yamatsu, A.; Matsumi, R. Isolation and Characterization of a Novel Poly(vinyl alcohol)-degrading Bacterium, Sphingopyxis sp. PVA3. Appl. Microbiol. Biotechnol. 2006, 72, 804-811.
-

- 186. Zhang, Y.; Li, Y. A New Strain, Streptomyces Venezuelae GY1, Producing a Poly(vinyl alcohol)-degrading Enzyme. World J. Microbiol. Biotechnol. 2006, 22, 625-628.
-

- 187. Monina, A.; Apryatina, K. Biodegradable Material Based on Starch-g-polyvinyl Acetate Copolymer with Bactericidal Properties. Polym. Bull. 2024, DOI:10.1007/S00289-024-05205-0.
-

- 188. Hesketh, A.; Cresswell, M. Microbial Biodegradation of Polyvinyl Acetate (PVAc) Emulsions. Int. Biodeterior. Biodegrad. 1995, 20, 288-296.
- 189. Qian, D.; Du, G. Isolation and Culture Characterization of a New Polyvinyl Alcohol-degrading Strain: Penicillium sp. WSH02-21. World J. Microbiol. Biotechnol. 2004, 20, 587-591.
-

- Polymer(Korea) 폴리머
- Frequency : Bimonthly(odd)
ISSN 0379-153X(Print)
ISSN 2234-8077(Online)
Abbr. Polym. Korea - 2023 Impact Factor : 0.4
- Indexed in SCIE
 This Article
This Article
-
2024; 48(4): 345-360
Published online Jul 25, 2024
- 10.7317/pk.2024.48.4.345
- Received on May 24, 2024
- Revised on Jun 2, 2024
- Accepted on Jun 7, 2024
 Services
Services
Shared
 Correspondence to
Correspondence to
- Dongyeop X. Oh** , and Dong-Ku Kang*, ***, ****
-
*Department of Chemistry, Incheon National University, Incheon 22012, Korea
**Department of Polymer Science and Engineering and Program in Environmental and Polymer Engineering, Inha University, Incheon 22212, Korea
***Bioplastic Research Center, Incheon National University, Incheon 22012, Korea
****Research Institute of Basic Sciences, Incheon National University, Incheon 22012, Korea - E-mail: d.oh@inha.ac.kr, dkkang@inu.ac.kr
- ORCID:
0000-0003-1261-6446, 0000-0003-3665-405X





















 Copyright(c) The Polymer Society of Korea. All right reserved.
Copyright(c) The Polymer Society of Korea. All right reserved.