- Synthesis and Characterization of KKRK Peptide-PAMAM Dendrimer G2 Conjugates as Gene Carriers
Department of Biochemistry, College of Natural Sciences, Chungnam National University, 99 Daehak-ro, Daejeon 34134, Korea
- 2세대의 PAMAM 덴드리머와 KKRK 펩티드를 접합한 고분자 소재의 합성 및 유전자 전달체 특성 연구
충남대학교 자연과학대학 생화학과
Reproduction, stored in a retrieval system, or transmitted in any form of any part of this publication is permitted only by written permission from the Polymer Society of Korea.
In this study, new hybrid polymers composed of polyamidoamine dendrimer generation 2 (PAMAM G2) and KKRK, KKRKH peptides derived from a nuclear localization sequence (NLS) were synthesized and characterized as gene carriers. Using Hep G2 and NIH3T3 cell lines, it was confirmed that the gene transfection efficiency was increased about 20- and 2700-fold in each cell compared to that of the native PAMAM G2. The main endocytosis mechanism of the polymer/plasmid DNA complex (polyplex) was proved to be macropinocytosis through endocytosis inhibition experiments using various inhibitors. It was confirmed that the polyplexes existed around the cell nucleus using a confocal microscope. In addition, it was observed that the polyplex showed active escape from endosomes using FACS analysis using acridine orange. As a result, KKRK-PAMAM G2 and KKRKH-PAMAM G2 show their potential to be one of efficient candidates as non-viral vectors.
본 연구에서, 2세대의 폴리도아민 덴드리머(PAMAM G2)와 nuclear localization sequence(NLS) 서열을 최적화한 KKRK, KKRKH 펩티드를 이용하여 새로운 하이브리드 고분자를 합성하였고 이들의 유전자 전달체로서의 특성을 확인하였다. Hep G2와 NIH3T3 세포주를 이용하여 유전자 형질변환 실험으로, 원래의 PAMAM G2 고분자와 비교하여 각 세포에서 20배, 2700배 이상 증가된 유전자 전달 효율을 확인하였다. 다양한 세포 내 이입 저해제들을 이용한 실험을 통해 본 고분자/플라스미드 DNA 복합체의 주된 엔도사이토시스 경로가 거대음작용 메카니즘임을 확인하였다. 공초점 현미경을 통해 플라스미드 DNA가 세포 핵 주변에 분포함을 확인하였다. 또한, FACS 실험으로 고분자/DNA 복합체인 폴리플렉스의 엔도좀의 탈출 효과가 활발함을 확인하였다. 본 연구를 통해 KKRK-PAMAM G2, KKRKH-PAMAM G2가 효율적인 비바이러스성 벡터의 후보군으로 활용될 수 있는 잠재적 가능성을 확인하였다.
New hybrid polymers composed of polyamidoamine (PAMAM) dendrimer generation 2 and KKRK, KKRKH peptides were synthesized and characterized as gene carriers. It was demonstrated that the gene transfection efficiency of the polymers was increased greatly in Hep G2 and NIH3T3 cell lines compared to that of the native PAMAM dendrimer.
Keywords: polyamidoamine dendrimer, nuclear localization sequence, cytotoxicity, endocytosis, transfection.
본 연구는 과학기술정보통신부의 차세대신약 기반기술 개발사업(NRF-2018M3A9B5024060, NRF-2018 M3A9B5024068)의 지원으로 수행되었습니다.
저자들은 이해상충이 없음을 선언합니다.
2019년 BIS Research에서 발표한 ‘Global cell and gene therapy market’에 따르면 세포 및 유전자치료제 시장의 규모는 2018년 6510만 달러이고 2025년까지 연평균 41%로 성장할 예정이다.1,2 암 및 만성질환 발생률 증가, 임상시험 건수 증가 등이 원인으로 파악되고 있으며 2020년 발표된 프로스트 앤설리번에 의하면 임상 단계에서 비바이러스성 벡터의 비중은 15%로 꾸준한 관심으로 연구를 진행하고 있다.3 하지만 신뢰 높은 벡터의 안정성과 가격, 맞춤형 서비스로 인해 높은 가격으로 측정되어 어려움을 가지고 있다.
비바이러스성 벡터 중 양이온성 고분자들은 폴리에틸렌이민(PEI), 폴리라이신(PLL), 덴드리머, 키토산, 젤라틴 등 다양한 화학적 구조와 물성을 갖으며, 이에 대한 연구가 지속적으로 이어지고 있다.4 그 중 폴리아미도아민(polyamidoamine, PAMAM) 덴드리머는 ethylenediamine(EDA) core에 EDA와 methyl acrylate(MA)가 반복되는 구형의 가지 구조를 갖고 있다.5 따라서, 덴드리머의 가지형 표면에는 다량의 아민기가 존재하여 양이온을 띄게 되고 정전기적인력에 의해 핵산과 나노사이즈의 폴리플렉스를 형성하며, 전달할 유전자를 다양한 효소분해로부터 보호할 수 있다.6 구조상 3차 아민들이 풍부하게 존재하여 엔도좀 완충 효과에 의해 pH가 서서히 떨어져 더 많은 이온들이 혼입하게 된다. 높아진 이온 농도는 삼투압의 증가를 유도하여 라이소좀의 성숙이 진행되기 전 엔도좀 막 구조가 붕괴되어 유전자 복합체가 탈출하게 된다. 이 가설은 ‘수소이온 스폰지 효과’로 세포 내부에 유전자 방출율을 높여 발현율이 높아진다고 알려져 있다.7
다양한 전략으로 PAMAM을 이용한 유전자 전달 벡터의 가능성을 높이는 연구들이 지속적으로 발표되었다.8,9 최근에는 PAMAM G2과 헤르페스 바이러스과에서 유래된 NLS인 RRILH, RRLHL을 도입하여 NIH3T3 세포주에서 PEI 보다 높은 유전자 형질전환 효율을 보이는 연구가 보고되었다.10 한편, 다른 전략으로 엔도좀 탈출을 증가시키기 위해서 카뎁신 B 효소에 특이적으로 절단되는 아미노산 서열(GFLG)을 도입하는 덴드리머 고분자를 합성하였고, Hela 세포주에서 높은 유전자 전달 효율을 확인하였다.11 또한, 인플루엔자 B 바이러스 핵단백질에서 유래된 NLS 서열인 KRTR, KRTRH를 PAMAM G3에 도입하여 합성하였고, 유전자 전달 효율이 A549 세포주에서는 PEI 보다 높고, Hep G2 세포주에서는 PEI와 유사한 값을 나타내는 연구 결과도 보고되었다.12
이러한 선행 논문 자료들을 참고하여, 본 연구에서는 낮은 세대의 폴리아미도아민 덴드리머와 유전자 전달 효율을 높일 수 있는 아미노산 서열을 합성하여 독성이 적고 유전자 전달 효율을 높일 수 있는 고분자를 합성하였다. 고전적인 NLS은 라이신과 아르기닌의 단일 cluster를 포함한 monopartite의 특성을 가지고 있다.13-15 해당서열을 가진 주된 NLS의 분석을 통해, SV40 바이러스에 있던 아미노산 서열, PKKKRKVE 서열과 NOSTRINβ에 존재하는 NLS 서열16 중 공통서열인 KKRK로 최적화하였고, 폴리아미도아민 덴드리머 2차에 펩티드 결합을 통해 합성하였다. 양이온성 고분자와 세포막의 인지질은 정전기적 인력으로 나노 크기의 구멍을 형성하는데,17 폴리아미도아민 덴드리머의 일차 아민의 수와 비례하여 유전자 전달 효율이 증가되지만 세포 독성을 유발할 수 있다. 따라서, 최적의 효율과 낮은 세포 독성을 위해 폴리아미도아민 덴드리머 2차를 선정하였다.
또한, 아미노산 서열 내에 히스티딘을 도입하였다. 히스티딘은 pKa가 6이고, 엔도좀에서 프로톤 스폰지 효과를 가지고 있어 폴리플렉스의 탈출을 촉진하여 세포독성을 감소시키는 것으로 알려져 있다.18 종합적으로 폴리아미도아민 덴드리머 2차와 히스티딘의 특성, 최적화한 NLS 아미노산 서열(KKRK)을 이용하여 KKRK-PAMAM G2, KKRKH-PAMAM G2의 물리화학적 특성과 새로운 유전자 전달체로서의 가능성을 확인하였다.
시약 및 재료. Polyamidoamine dendrimer ethylenediamine core generation 2 solution(PAMAM G2), N,N-dimethylform- amide(DMF), Fmoc-Lys(Boc)-OH, ethidium bromide(EtBr), piperidine, triisopropylsilane(TIS), trifluoroaceticacid(TFA), diisopropylethylamine(DIPEA), dimethylsulfoxide(DMSO), Deuterium oxide(D2O), acetonitrile(99.9%), chlorpromazine hydrochloride, methyl-β-cyclodextrin, nocodazole, genistein 는 Sigma-aldrich(St. Louis, MO, USA)에서 구매하였다. Fmoc-Arg(Pbf)-OH, ANASPEC(Fremont, CA, USA)에서 구매하였다. N-hydroxybenzotrizole(HOBt), 2-(1H-benzotriazolel-yl)-1,1,3,3-tetramethyluronium(HBTU), Fmoc-His(Trt)-OH는 Novabiochem(Darmstadt, Germany) 사에서 구입하였다. Ethyl ether, 99%는 Junsei(Nihonbashi-honcho, Chuoku, Tokyo, Japan)에서 구매하였다.
Dulbecco's modified eagle's medium(DMEM), Dulbecco's phosphate buffered saline(DPBS), fetal bovine serum(FBS), WELGENE(Daegu, South Korea)에서 구매하였다. Trypsin 0.25%(1X) solution은 HyClone(Logan, UT, USA)에서 구매하였다.
Reporter Lysis 5X buffer, luciferase assay system는 Promega(Madison, Wisconsin, USA)에서 구매하였다.
100×antibiotic-antimycotic reagent, bisbenzimide(Hoechst 33342), Alexa 546 Nucleic Acid labeling kit, Micro BCATM Protein Assay Kit, acridine orange(Molecular Probes)는 Thermo Fisher Scientific(Carlsbad, CA, USA)에서 구매하였다.
WST-8 Cell Viability Assay Kit는 BIOMAX(Seoul, South Korea) 사에서 구매하였다.
세포주. Hep G2(human hepatocellular carcinoma cell line) 세포주와 NIH3T3(mouse embryonic fibroblast cell line) 세포주는 10% FBS와 15 antibiotic-antimycotic reagent가 포함된 DMEM을 이용해 37 oC, 5% CO2, 95% 유지된 배양기에서 배양하였다.
KKRK, KKRKH-G2 합성. 최근에 발표된 논문과 같은 방법을 사용하였다.19 간략히 말하면, 액체상 펩티드 합성법을 이용해서 PAMAM Generation 2(G2)와 Fmoc-lys(boc)-OH, HOBt, HBTU, DIPEA는 1:4:4:4:8의 몰비율로 DMF에서 18시간 동안 43 oC에서 반응하였다. 그 후, 합성된 물질은 차가운 디에틸에테르로 침전하고, 씻어준다. 침전된 합성물을 질소가스로 건조시킨 후 7:3(v/v)의 DMF/piperidine 혼합물에 용해시켜 암조건 하에서 2시간 동안 Fmoc 보호기 제거를 진행하였다. 이후에 다음 아미노산 서열들을 동일한 방법으로 펩티드 합성을 진행한다. 아르기닌의 Pbf, 히스티딘의 Trt, 라이신의 Boc 보호기 제거를 위해 TFA/TIS/3차 증류수(부피비 95:2.5:2.5)에 용해시키고 7시간 동안 반응시켰다. 이후에 디에틸에테르로 침전 후 씻어준 다음에 3차 증류수에 용해시켜 투석막(MWCO 3500, Spectra/Por)에 옮긴 후 24시간 동안 투석을 진행하였다. 투석 후 동결건조 진행하였고, 합성률을 확인하기 위해 600 MHz 핵자기공명분석법(NMR)으로 분석하였다.
HPLC(고성능 액체 크로마토그래피) 순도 분석. G2, KKRKH-G2는 2000 ppm 농도로, KKRK-G2는 1000 ppm 농도로 진행하였다. 합성물의 10 mL를 HPLC에 사용하였고, 컬럼은 C18(5 mm, 2.1×100 mm)를 이용하여 검출기 파장은 210 nm, 컬럼온도는 25 oC, 속도는 0.4 mL/min 조건에서 10분간 0.1% 트리플루오로아세트산이 들어있는 물과 아세토니트릴을 95:5에서 75:25의 구배로 분석하였다.20
KKRK, KKRKH-G2와 플라스미드 DNA의 폴리플렉스 형성 분석. 0.5 mg pDNA(pCN-luc)와 폴리머를 중량비에 따른 결합력을 알아보기 위해서 전기영동하였다. HEPES buffer (125 mM, pH 7.4) 상에서 중량비대로 준비한 뒤, 30분 동안 상온에서 complex시킨다. 0.7% 아가로스겔에 EtBr 0.5 mg/μL를 처리해서 100 V에서 15분 동안 전기영동을 수행하였다.
입도분석기를 이용한 폴리플렉스의 크기 및 제타전위 측정. 폴리머와 pDNA의 폴리플렉스 중 세포 내 유전자 전달효율과 독성을 고려한 최적의 중량비를 이용하여 상온에서 30분 동안 complex시킨다. 그 이후 각각 3차 증류수를 첨가하여 Zetasizer Nano ZS(Malvern Instruments Ltd., Malvern, UK)로 측정하였다.
세포 내 독성 실험. HepG2와 NIH3T3 세포주에서 KKRK, KKRKH-G2의 농도에 따른 독성을 확인하기 위해 WST-8 assay(Biomax, Quanti-Max)를 수행하였다. 각 세포를 96 well plate에 18,000 세포/well 로 분주한 뒤, 37 oC, 5% CO2, 95% 습도조건에서 배양하였다. 18시간 후, 다양한 농도로 준비된 PEI 25 kDa, G2, KKRK-G2, KKRKH-G2를 세포에 처리하고 24시간 동안 동일 조건에서 배양하였다. 다양한 폴리머들의 처리시간 24시간에 도달하기 2시간 전에 Ez-Cytox reagent WST-8 시약을 각 well 당 10 μL씩 처리한 후, 2시간 동안 반응시켰다. 이후, ELIZA(VERSAmax, Molecular Devices, Sunnyvale, CA, USA)로 450 nm의 흡광도에서 측정하였다.
세포 내 유전자 전달 효율 실험. KKRK, KKRKH-G2의 세포 내 유전자 전달 효율을 확인하기 위해 HepG2와 NIH3T3 세포주에서 진행하였다. 각 세포를 96 well plate에 18000 세포/well로 분주한 뒤, 37 oC, 5% CO2, 95% 습도조건에서 배양하였다. Complex test와 동일한 방식의 중량비로 세포 배양액을 첨가하여 30 μL의 폴리플렉스를 만들고, 세포가 분주된 후 18시간에 도달할 때 세포에 처리하였다. 폴리플렉스가 처리된 후 24시간이 되었을 때, 세포 배양액을 제거하고 DPBS로 씻어준 후 Reporter Lysis Buffer(Promega)를 상온에 30분간 반응시켜 세포를 용해시킨다. 13200 rpm, 10분간 원심분리기를 이용해서 상층액을 분리시키고, 상층액을 사용하여 Micro BCATM Protein Assay(Pierce, Rockford, IL, USA)를 이용해서 생성된 단백질을 정량하고 Lumat LB 9507(Berthold Technology, Bad Wildbad, Germany)로 luciferin agent (Promega)를 처리하여 루시페라아제 활성을 측정하였다. 최종적으로 RLU/μg protein의 단위로 나타내었다.
세포 내 이입 저해 메카니즘 실험. 폴릭플렉스가 어떠한 종류의 세포 내 이입을 통해 들어가는지 알아보기 위해 실험을 진행하였다. NIH3T3 세포주를 96 well plate에 18000 세포/well 로 분주한 뒤, 37 oC, 5% CO2, 95% 습도조건에서 24시간 동안 배양하였다. Complex test와 동일한 방식의 중량비로 세포 배양액을 첨가하여 30 μL의 폴리플렉스를 만들고, 폴리플렉스 처리 30분 전 세포 내 이입 저해제들을 최종 농도 7.5 μM 클로르프로마진, 20 μM methyl-β-cyclodextrin, 10 μM 노코다졸, 37 μM 제니스테인으로 세포 배양액과 함께 기존 세포 배양액에서 바꿔주었다.21 폴리플렉스가 처리된 후 6시간이 지나면, 기존의 세포 배양액을 제거하고 동일한 농도의 세포 내 이입 저해제가 포함된 세포 배양액으로 변경하였다. 세포 내 이입 저해제에 노출된 후 12시간이 지나면 세포 내 유전자 전달 효율 실험과 동일한 방법으로 세포를 용해시키고 상층액을 수득하여 루시페라아제 활성을 비교하였다.
공초점 형광현미경 실험. 세포 내로 pDNA가 잘 들어갔는지 확인하기 위해서 공초점 형광현미경을 사용하였다. pDNA는 Alexa 546 Nucleic Acid Labeling Kit의 protocol에 명시된 것과 동일하게 형광표지 하였다. NIH3T3 세포주를 1 μ-Slide 8 well(Ibidi)에 30000 세포/well 분주한 뒤, 37 oC, 5% CO2, 95% 습도조건에서 24시간 동안 배양한다. 그 이후 형광표지된 pDNA와 PEI 25 kDa, G2, KKRK-G2, KKRKH- G2의 폴리플렉스를 세포에 처리해준다. 24시간 동안 배양한 후, 공초점 현미경으로 분석하기 30분 전에 세포핵을 Hoechst 33342으로 10분간 반응시켜 염색하고 Zeiss LSM 5 live scanning microscope(Carl Zeiss, Jena, Germany)를 통해 관찰하였다.
유세포 분석(Flow Cytometry) 실험. 세포 내 폴리플렉스가 유전자 전달을 한 후, 엔도좀 탈출을 위해 라이소좀으로 변화되어 산성을 나타내는 것을 확인하기 위해 아크리딘 오렌지(acridine orange, AO)를 이용하여 실험을 진행하였다. NIH3T3 세포주를 6 well plate에 150000 개/well로 분주한 뒤, 37 oC, 5% CO2, 95% 습도조건에서 24시간 동안 배양하였다. 0.5 μg pDNA(pCN-luc)와 KKRK, KKRKH-G2를 1:8 중량비로 세포배양액 상에서 600 μL의 complex를 상온에서 30분간 형성하였다. 해당 폴리플렉스를 배양 후 24시간이 되었을 때, 세포에 처리하였다. 폴리플렉스의 처리시간이 24시간이 되었을 때, 세포를 떼어내서 3000 rpm, 3분간 원심분리시켜 상층액을 수득하였다. 세포를 한 번 더 씻어, 동일한 조건으로 원심분리를 진행하고 AO의 농도가 2.5 μM가 되도록 처리하여 염색하였다. 유세포분석기(Becton Dickinson, FACS Canto II)를 이용해서 FITC, Texas Red 파장대에서 10000개의 세포를 분석하였다.22
통계 처리. 실험 결과 데이터의 통계 처리는 GraphPad사의 Prism 5 프로그램을 사용하여 Student’s t-test를 수행하였다(*p<0.05, **p<0.01 and ***p<0.001).
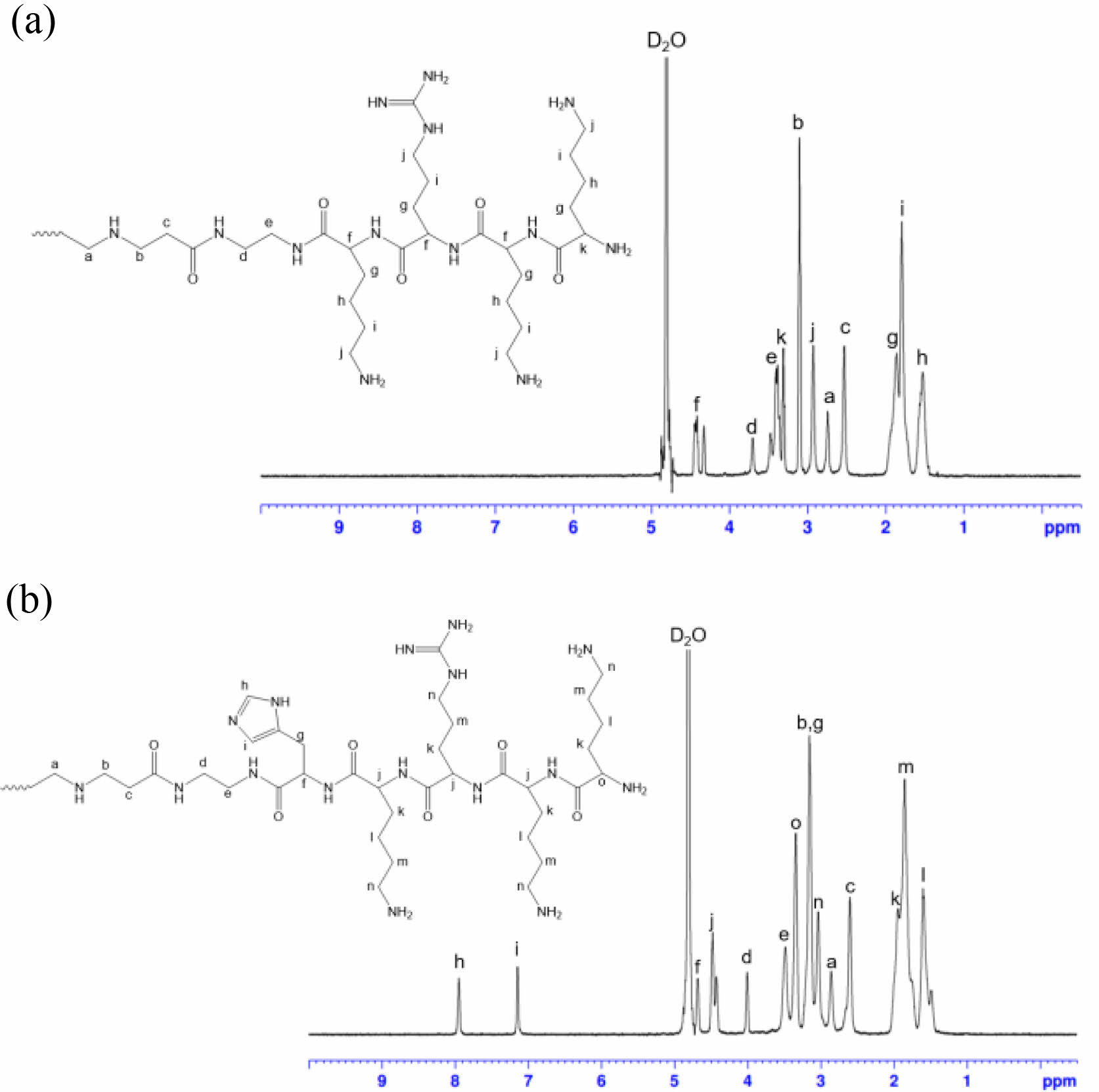
KKRK, KKRKH-PAMAM Generation 2(G2) 합성. 본 연구에서 제작한 KKRK-G2는 PAMAM에 라이신, 아르기닌 순으로 차례로 합성하였고, KKRKH-G2는 히스티딘, 라이신, 아르기닌 순으로 합성을 하였다. 산화 중수소(D2O)에 녹인 합성물을 600 MHz 핵자기공명분석법(NMR)으로 분석한 결과, KKRK-G2의 합성률은 라이신, 아르기닌이 각각 99%였고, 최종 99%이상의 합성률을 보였다. KKRKH-G2의 합성률은 라이신, 아르기닌은 99%, 히스티딘은 92%로 최종 전체 합성률은 92% 이상으로 확인하였다. NMR 스펙트럼 분석은 다음과 같다(Figure 1).
KKRK-G2: δ(ppm) 1.545(라이신 -CHCH2CH2CH2CH2NH2-), 1.789(라이신 -CHCH2CH2CH2CH2NH2-, 아르기닌 -CHCH2- CH2CH2NH-), 1.856(라이신 -CHCH2CH2CH2CH2NH2-, 아르기닌 -CHCH2CH2CH2NH-), 2.526(PAMAM -NCH2CH2CO-), 2.738(PAMAM -CONHCH2CH2N-), 2.926(라이신 -CHCH2- CH2CH2CH2NH2-, 아르기닌 -CHCH2CH2CH2NH-), 3.089 (PAMAM -NCH2CH2CO-), 3.306(라이신 -NH2CHCH2CH2- CH2CH2NH2-), 3.382(PAMAM -NCH2CH2NCO2-), 3.7(PAMAM -NCH2CH2NCO-), 4.414(라이신 -CHCH2CH2CH2CH2NH2-, 아르기닌 -CHCH2CH2CH2NH-).
KKRKH-G2: 1.589(라이신 -CHCH2CH2CH2CH2NH2-), 1.852 (라이신 -CHCH2CH2CH2CH2NH2-, 아르기닌 -CHCH2CH2- CH2NH-), 1.939(라이신 -CHCH2CH2CH2CH2NH2-, 아르기닌 -CHCH2CH2CH2NH-), 2.604(PAMAM -NCH2CH2CO-), 2.858 (PAMAM -CONHCH2CH2N-), 3.035(라이신 -CHCH2CH2- CH2CH2NH2-, 아르기닌 -CHCH2CH2CH2NH-), 3.154(PAMAM -NCH2CH2CO-, 히스티딘 -HCCH2CCHNCHNH-), 3.340(라이신 -NH2CHCH2CH2CH2CH2NH2-), 3.484(PAMAM -NCH2- CH2NCO-), 4.009(PAMAM -NCH2CH2NCO-), 4.479(라이신 -CHCH2CH2CH2CH2NH2-, 아르기닌 -CHCH2CH2CH2NH-), 4.679(히스티딘 -HCCH2CCHNCHNH-), 7.145(히스티딘 -HCCH2CCHNCHNH-), 7.95(히스티딘 -HCCH2CCHNCHNH-).
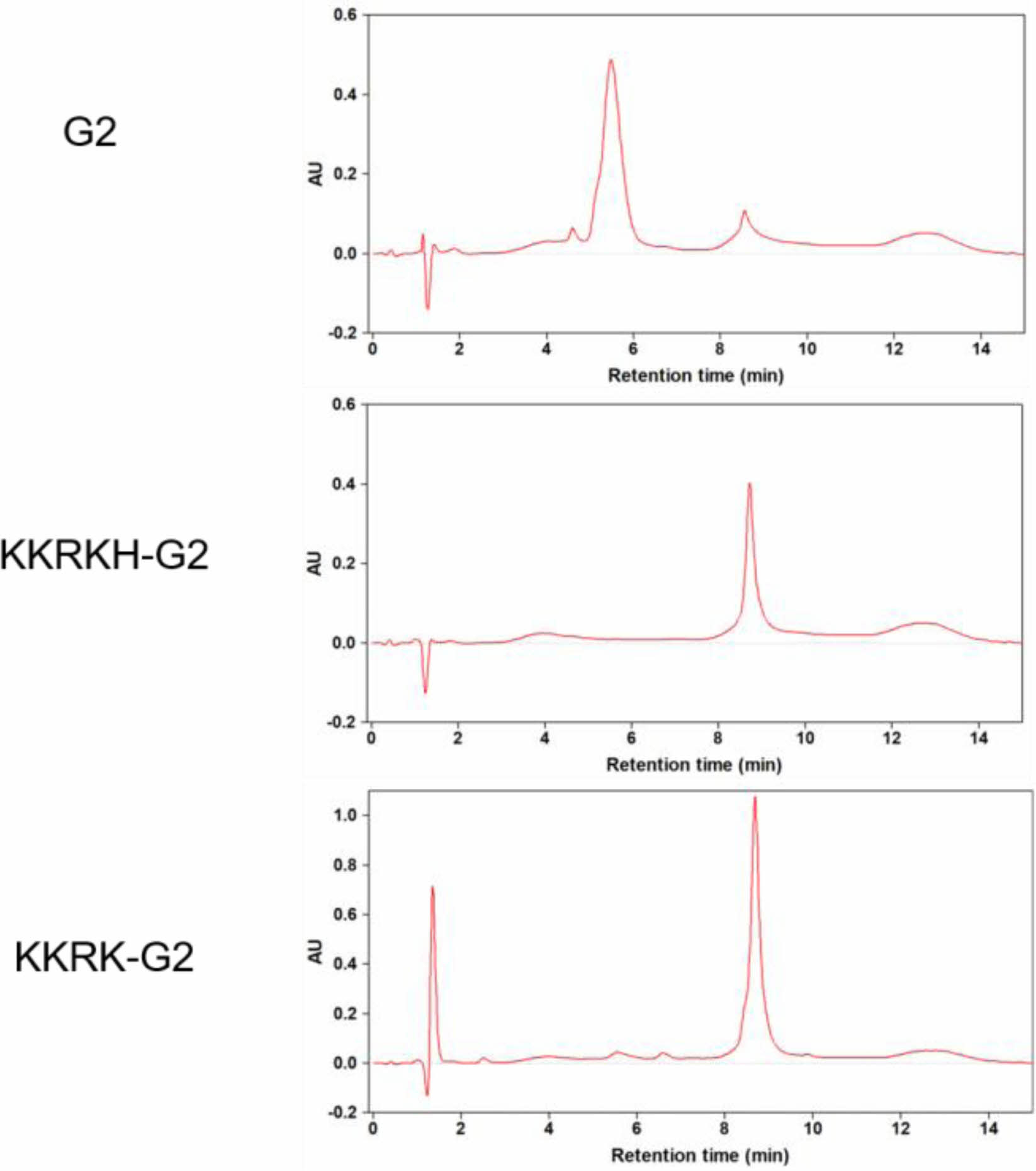
HPLC(고성능 액체 크로마토그래피) 순도 분석. NMR분석을 통해 90% 이상의 합성률을 확인하였고, HPLC를 이용해서 순도 분석을 진행하였다. 실험 조건에 따라 아미노산이 합성되어 분자량이 커진 KKRK, KKRKH-G2는 기준인 G2의 머무름 시간인 5분보다 늦은 9분에 나타나는 것을 확인할 수 있었다. 또한, 가장 큰 피크로 나타난 것으로 보아 순도 높은 합성이 되었다는 것을 확인할 수 있다(Figure 2).
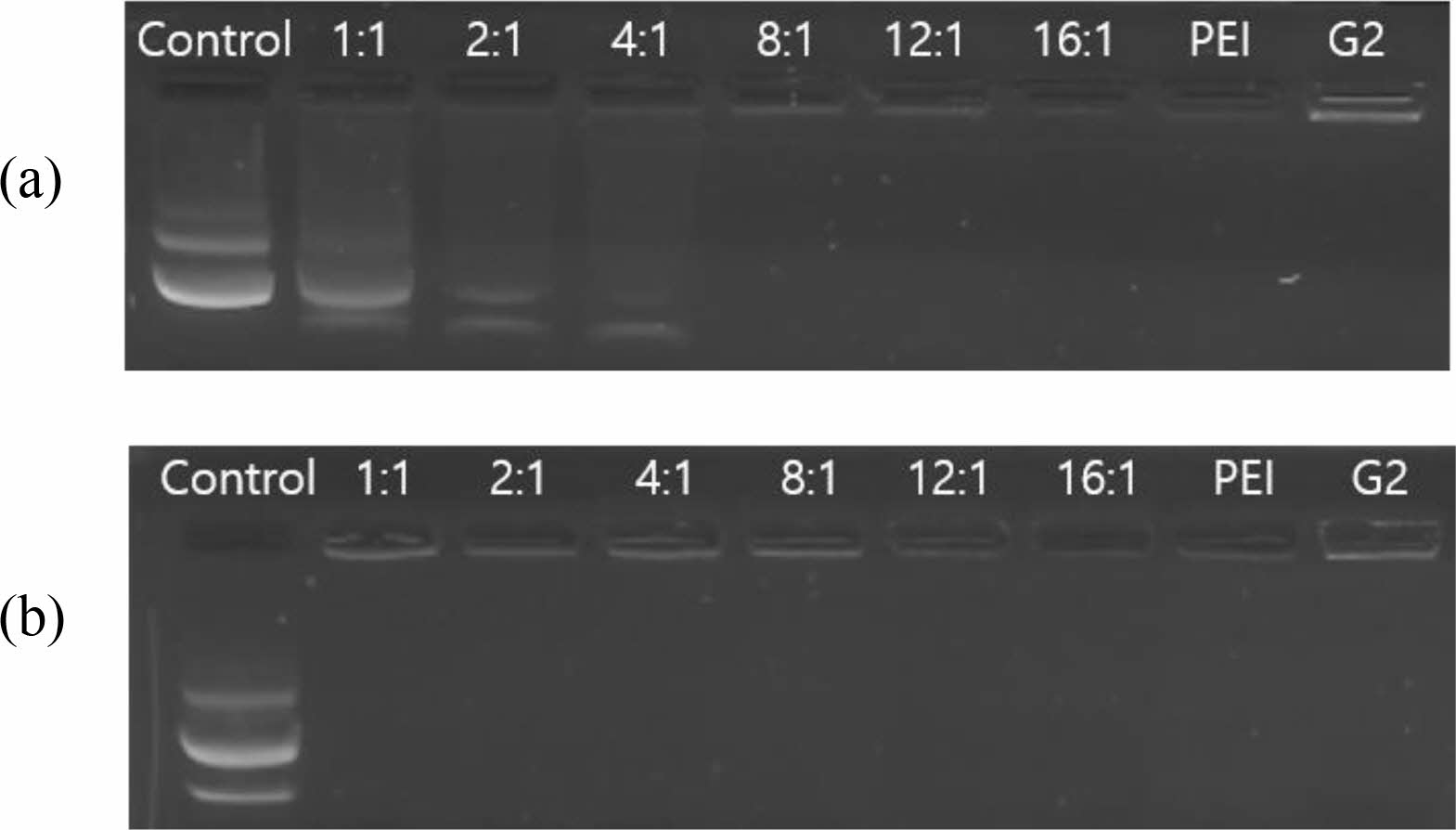
플라스미드 DNA의 Complex Test와 크기 및 제타전위 측정. 다양한 중량비로 합성물과 플라스미드 DNA(pDNA)이 폴리플렉스를 형성하는지 확인하기 위해서 전기영동을 진행하였다. KKRKH-G2는 1:1 부터 pDNA의 밴드가 완벽하게 복합체를 형성한 것을 확인하였다. KKRK-G2는 4:1부터 pDNA의 밴드가 줄어들며, 8:1은 완벽하게 복합체가 형성됨을 확인하였다(Figure 3).
전기영동을 통한 complex test 통해서 각 합성물들이 8:1에서 완전한 폴리플렉스를 만드는 것을 확인하였다. Zetasizer로 분석한 결과, 실제로 KKRKH-G2는 +33.8 mV, KKRK-G2는 +26.13 mV가 나타나는 것을 확인하였다. 세포 내로 유전자가 이입되기 위해서 폴리플렉스는 300 nm 이하의 나노사이즈를 가지는 것이 중요한데, 해당 폴리플렉스는 178, 176 nm를 나타나는 것을 확인하였다(Table 1).
결과적으로 KKRK-G2, KKRKH-G2의 충분한 양전하 특성은 Zetasizer 분석으로 증명하였고, pDNA의 인과 정전기적 인력을 통해 폴리플렉스가 형성됨을 전기영동을 이용하여 확인하였다.
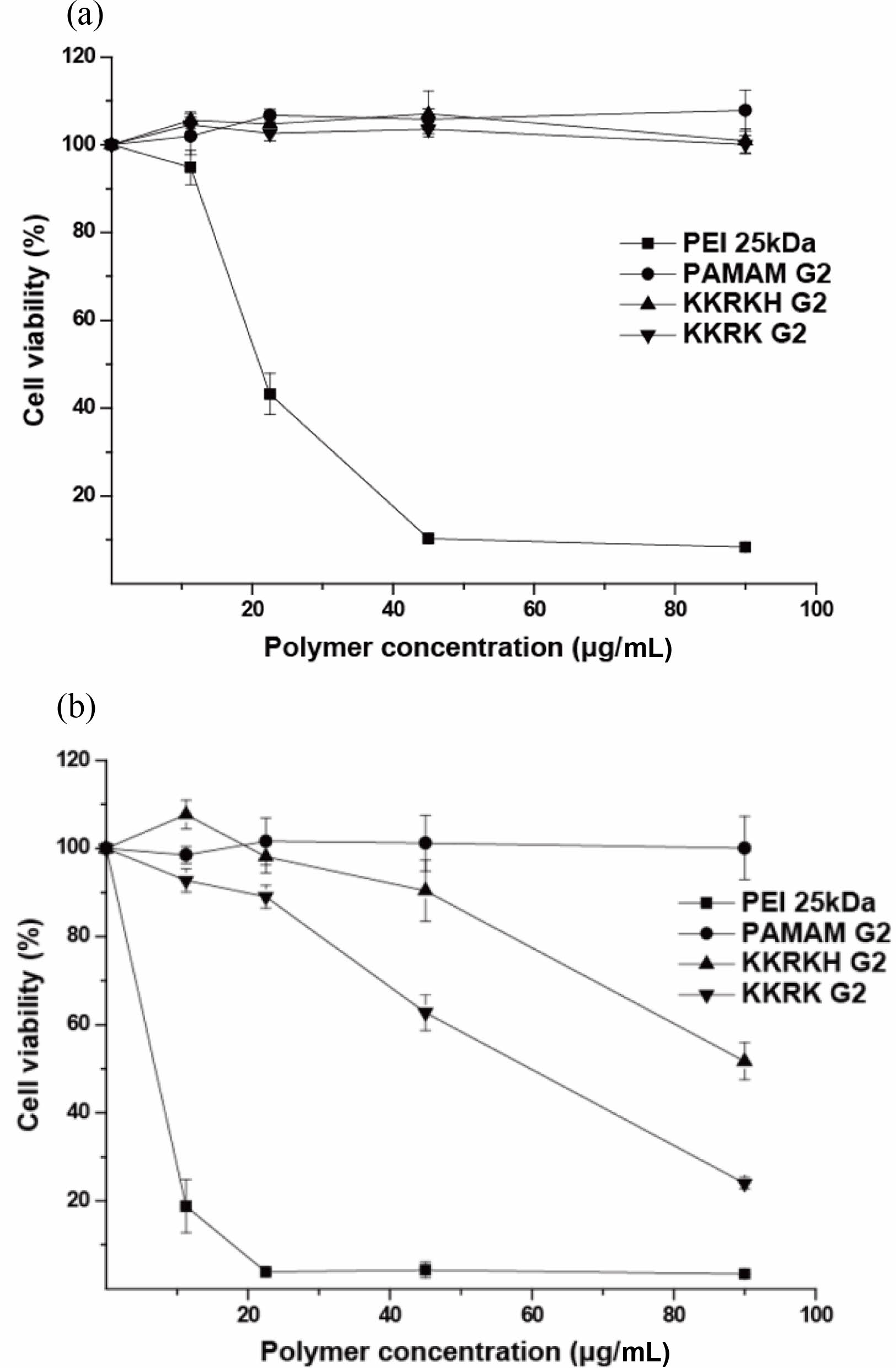
세포 내 독성 실험. 세포주에 따른 KKRK-G2, KKRKH-G2의 독성을 평가하기 위해서 WST assay를 진행하였다.
살아있는 세포는 미토콘드리아 수소이탈효소에 의해 환원되어 formazan을 형성하게 되고 죽은 세포는 효소활성이 사라져 formazan의 흡광도에 따라 세포독성을 분석할 수 있다. PEI, G2, KKRK-G2, KKRKH-G2는 최종 농도가 90, 45, 22.5, 11.25 μg/mL되도록 세포에 처리하였다. HepG2 세포주에서는 세포 독성이 높다고 알려진 PEI 25 kD는 매우 높은 독성을 나타낸 것을 확인하였고, 해당 농도에서 G2, KKRK-G2, KKRKH-G2가 독성이 없음을 확인할 수 있었다(Figure 4). NIH3T3 세포주에서도 PEI 25 kD의 독성을 확인하였고, KKRK-G2는 90 μg/mL에서 24%, 45 μg/mL에서 8%의 세포 생존율이 나타났다. 양전하를 가진 나노입자는 일반적으로 음전하를 가진 입자보다 세포 내로 유입이 더 많아서 세포 독성에 영향을 주는 것으로 알려져 있다.23 또한, 양이온성 고분자는 음이온성 고분자에 비해 세포막의 안정성을 방해하고, 사이클린(D,E)의 발현을 감소시킨다. 이는 세포 주기 중 G1이 지연되고, 세포분열을 저해하게 되어 독성을 나타내는 경우도 보고되었다.24 KKRK, KKRKH-G2는 세포주별로 독성 차이가 나타났는데, 세포의 민감도에 의한 세포막 손상 정도의 차이라고 사료된다. 보고에 따르면, 양이온성 물질을 이용한 세포실험에서 NIH 3T3의 IC50 값은 4 ppm이고 HepG2의 IC50는 97 ppm으로 같은 물질의 독성 정도가 24배 차이가 나는 것을 볼 때, NIH 3T3 세포의 양이온성 물질에 대한 민감도가 Hep G2보다 더 높은 것에 기인하는 것으로 생각된다.25 KKRK, KKRKH-G2는 덴드리머 형태의 구조에 양전하를 가지는 아미노산 서열들이 연속해서 4개가 붙어있어 세포 골격 속 음전하의 단백질들(액틴, 베타 튜블린 등)과 반응하여 세포막을 방해시켜 손상되어 독성을 나타내는 것으로 사료된다.24,26,27 하지만, KKRKH-G2는 90 g/mL에서 51%의 세포 생존율을 보여 고농도에서는 독성이 나타나지만 KKRK-G2보다 생존율이 6배 이상 증가하는 것을 확인하였다. 최근에 발표된 논문에서 제시하는 바와 같이 다수의 히스티딘 잔기 도입을 통해 세포 내 활성 산소종을 제거하여 독성을 낮추는 효과의 가능성을 확인하였다.28
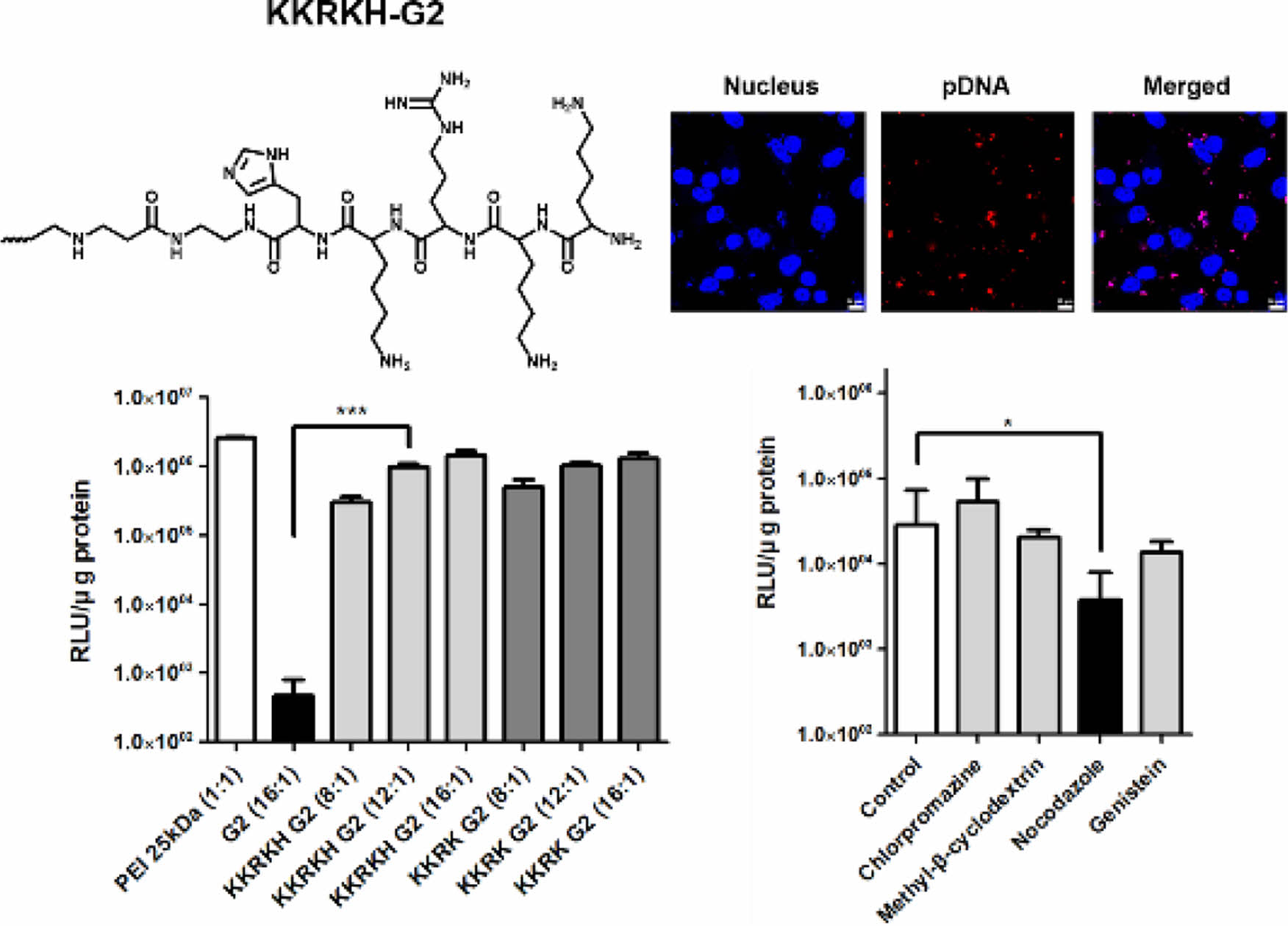
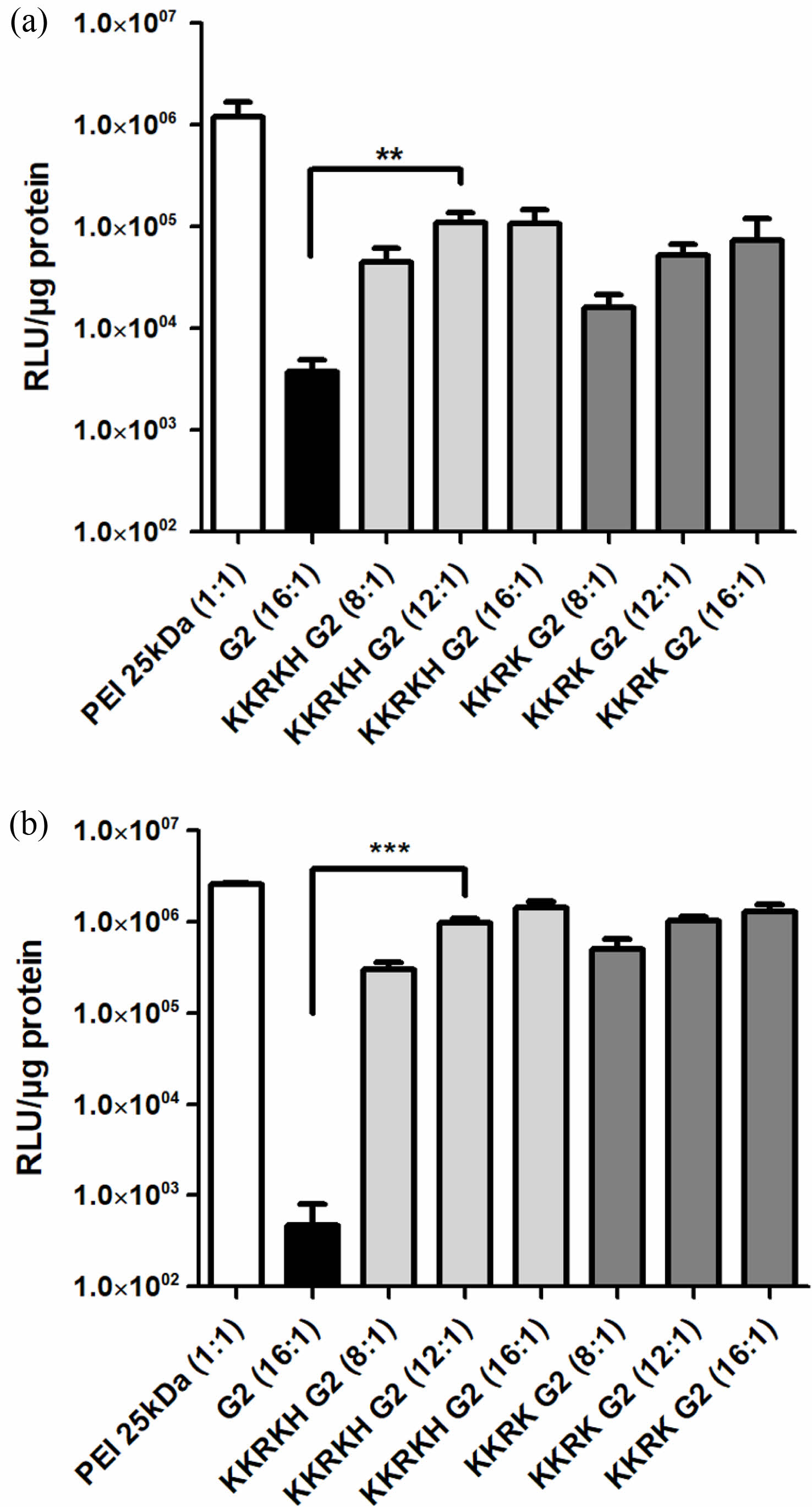
세포 내 유전자 전달 효율 실험. 핵 위치 서열의 특성을 가진 KKRK, KKRKH-G2의 세포 내 유전자 전달 효율을 확인하기 위해서 Hep G2와 NIH3T3을 사용하였다. 두가지 세포주에서 PEI 25 kD는 플라스미드 DNA와 중량비 1:1의 폴리플렉스를 형성하였고, 유전자 효율이 높다고 보고된 결과와 일치함을 확인하였다.8
세포 내 유전자 전달 효율은 아르기닌과 라이신, 히스티딘을 도입한 것이 아미노산을 도입하지 않은 PAMAM 덴드리머보다 증가한 것을 확인하였다(Figure 5). Hep G2 세포주의 경우 동일한 중량비에서 G2보다 KKRKH-G2는 29배, KKRK-G2는 20배 증가하였고, NIH3T3 세포주에서는 동일한 중량비에서 G2보다 KKRKH-G2는 3011배, KKRK-G2는 2779배 증가하였다. PEI는 43 Da마다 1개의 양이온이 존재하여, 하전 밀도가 매우 높은 양이온성 고분자이다. KKRK-G2는 186 Da마다 1개, KKRKH-G2는 220 Da마다 1개의 양이온이 존재한다. 그러므로 PEI보다 하전 밀도가 상대적으로 더 낮고 플라스미드 DNA의 응집력 또한 차이가 난다고 볼 수 있다. 따라서, 본 연구에서 수행한 두 가지 세포주에서 PEI와 유사하거나 낮은 유전자 전달 효율을 나타냈다고 생각한다. 하지만, 두 종류의 세포주에서 동일하게 순수한 G2 덴드리머 폴리플렉스의 중량비보다 낮은 비율에서 KKRK-G2 및 KKRKH-G2 폴리플렉스들의 유전자 전달 효율이 급격하게 증가되어 NLS에 의한 유전자 전달 효율이 통계적으로 유의미하게 증가하였음을 확인하였다. 이는 폴리플렉스상의 양이온성 펩티드로 인한 세포투과성이 증가되었고, 또한 NLS 서열 효과에 의한 핵 타켓팅 효율의 증가에 기인한다고 사료된다.
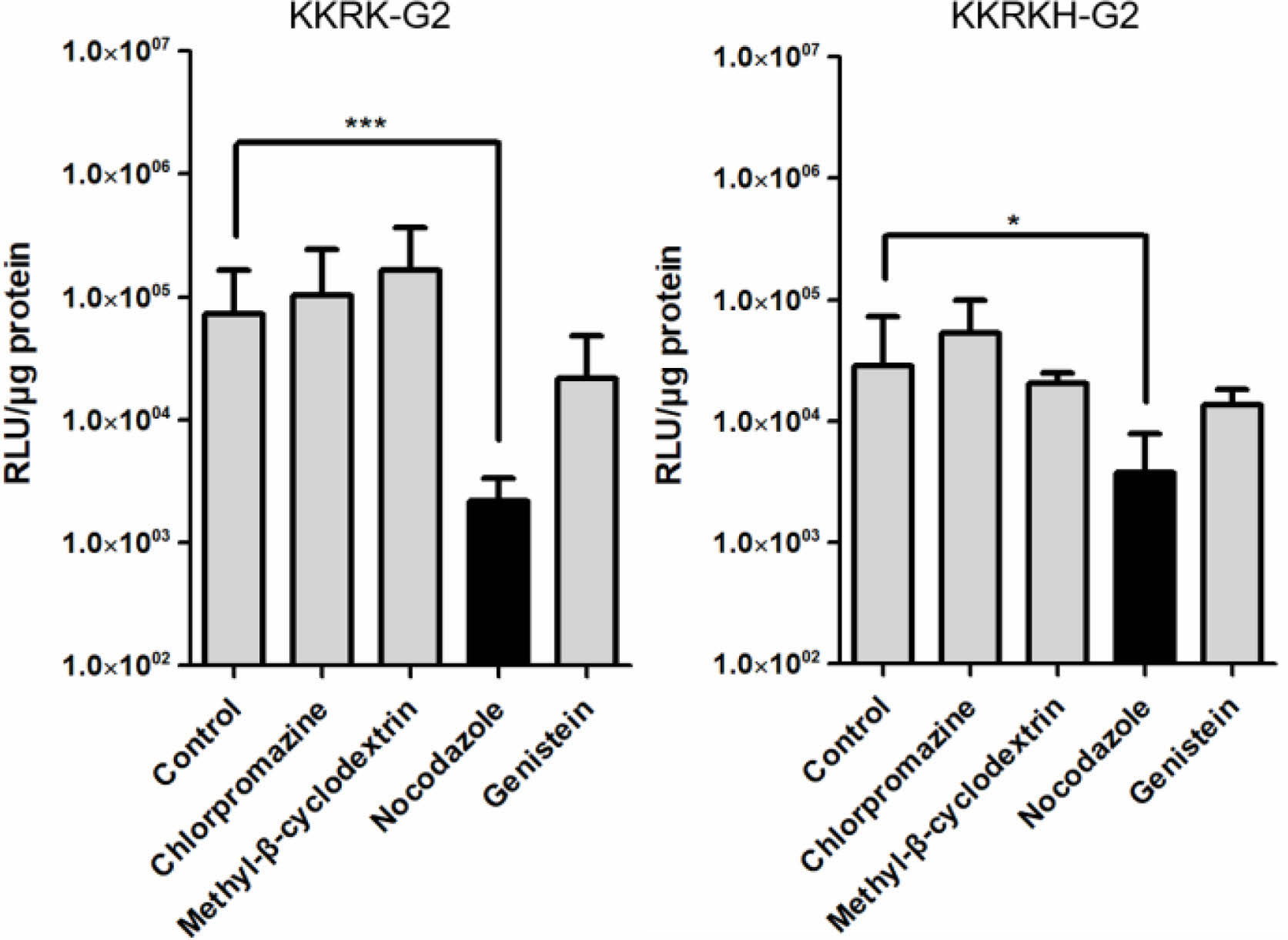
세포 내 이입 저해 메카니즘 실험. KKRK, KKRKH-G2/pDNA 폴리플렉스가 어떻게 세포 내로 이입되는지 다양한 억제제를 사용하여 진행하였다. 각 억제제의 원리를 간단히 나열하면 다음과 같다. 클로르프로마진은 클라트린 매개 세포 내 이입과정 억제제이고, methyl-β-cyclodextrin은 카베올리와 지질들이 인터널리제이션되는 클라트린 비매개(지질 뗏목 경로) 세포 내 이입과정, 노코다졸은 거대음작용, 제니스테인은 Src 타이로신 카이네이즈를 억제하는 클라트린 비매개 세포 내 이입 억제제다.21,29
PAMAM은 세포에 따른 다양한 세포 내 이입과정들이 보고되어 왔다.30,31 억제제를 사용하지 않은 대조군과 비교했을 때, 같은 농도에서 KKRK-G2, KKRKH-G2들은 노코다졸에 의한 유전자 전달 효율이 급격히 감소하는 것을 확인하였다(Figure 6). 억제제 효과가 통계적으로 유의미함을 보여주었기에, KKRK-G2, KKRKH-G2들은 거대음작용을 통한 세포 내 이입이 진행될 수 있다는 가능성을 입증하였다.
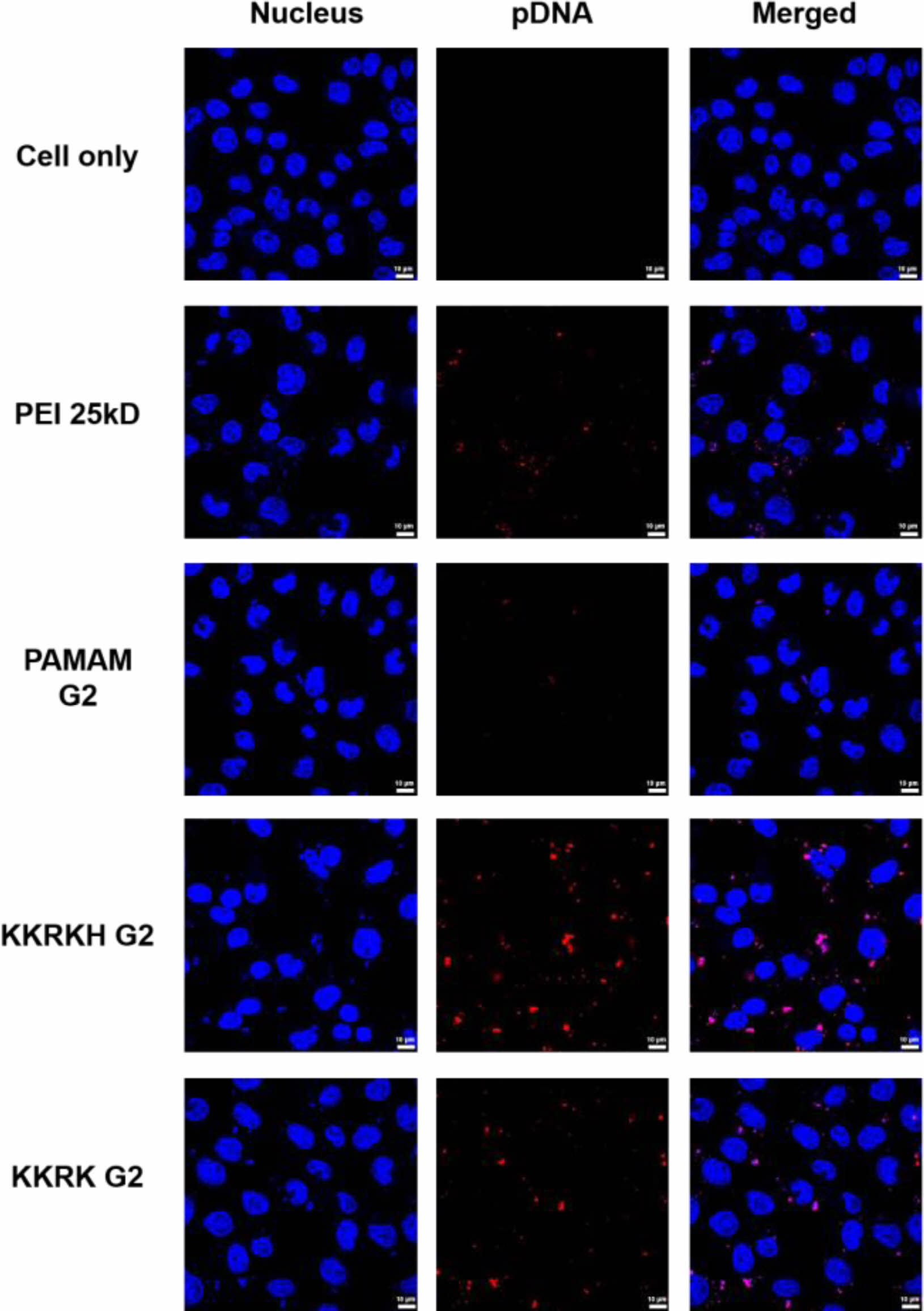
공초점 형광현미경 실험. 세포 내 유전자 전달 효율이 증가되려면 폴리플렉스들이 세포막 내로 이입되고 라이소좀 성숙에서 탈출하여 DNA가 핵 내부로 들어가 유전자 발현이 잘 되어야 한다.
실제 이미지 상에서 유전자가 세포 핵주변으로 전달되는지 확인하기 위해서 실험을 진행하였다. 대표적으로 NIH3T3 세포주 내 유전자 전달 효율이 높고 세포 독성이 적은 KKRKH-G2 폴리플렉스를 사용하였고, 중량비는 KKRKH-G2와 플라스미드 DNA는 8:1로 진행하였다.
세포 내 유전자 전달 효율과 유사한 결과로 PAMAM G2보다 KKRK-G2, KKRKH-G2들이 세포 핵 근처에 플라스미드 DNA가 존재하는 것을 볼 수 있다(Figure 7). 그 중 KKRKH-G2는 KKRK-G2보다 유전자 전달 효율이 높았는데, 이미지 상에서 더 많은 플라스미드 DNA를 전달하였다.
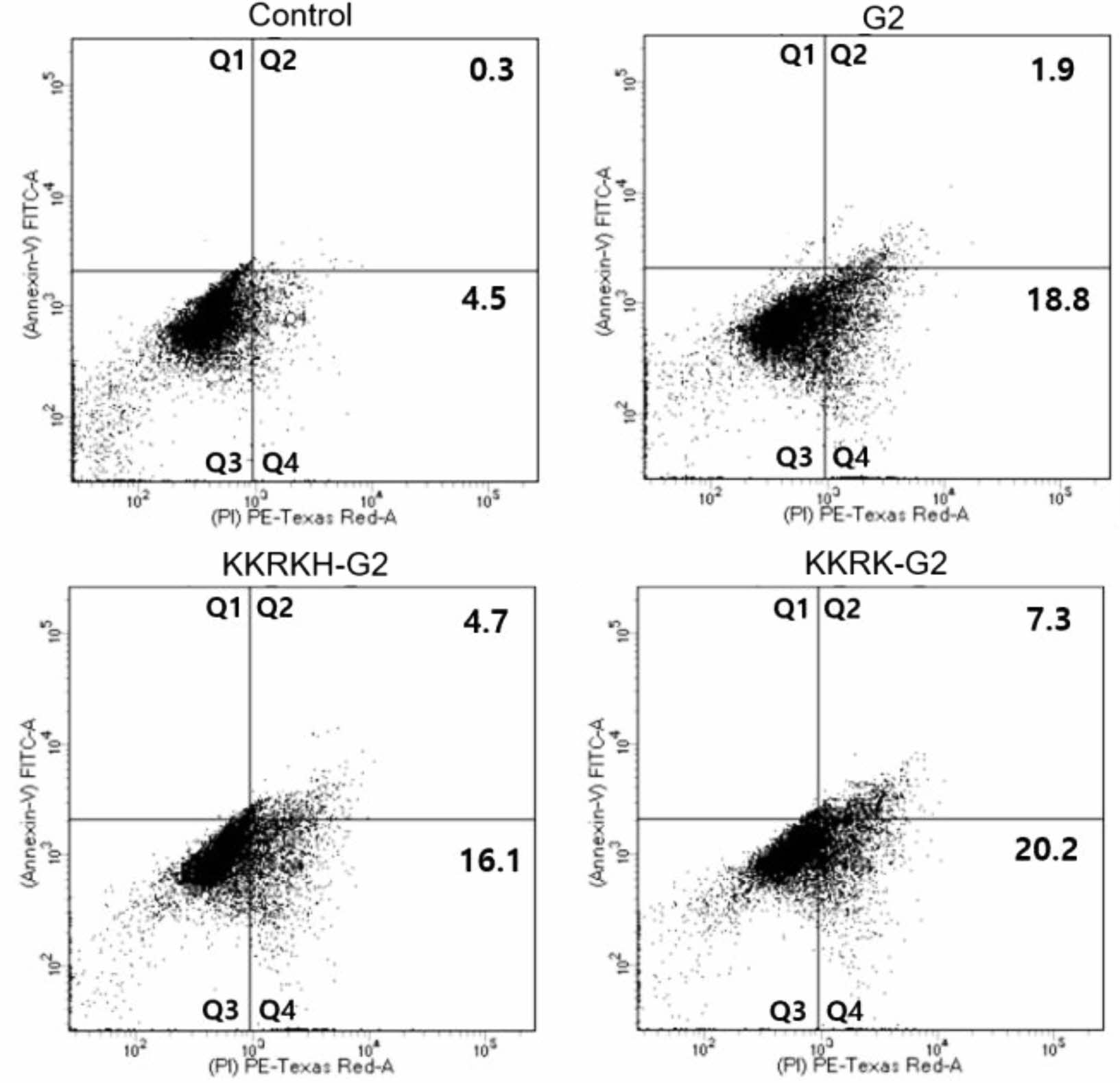
폴리플렉스의 엔도좀 탈출 및 세포질 분포 효능 분석. 아크리딘 오렌지는 세포 내에서 이중나선 DNA의 인과 반응하여 양성자화된 AO가 530 nm에서 초록색의 형광을 나타낸다. 하지만 산성화된 환경에서는 양성자화된 아크리딘 오렌지가 응집되어 630 nm에서 최대 형광을 나타내 빨강-오렌지 색을 나타낸다고 알려져 있다.32 폴리플렉스가 세포 내로 이입되어 라이소좀으로 성숙되가는 과정에서 아크리딘 오렌지는 빨강-오렌지 색의 형광을 나타나게 되지만 엔도좀을 탈출하면 기존의 초록색과 빨간색이 섞인 노란색 형광을 띄게 된다.33 폴리플렉스가 NIH3T3 세포주 내로 폴리플렉스에 노출된지 24시간이 지난 후 유세포분석기로 분석한 결과, G2, KKRK-G2, KKRKH-G2을 처리한 세포들은 Q4부분에서 아무것도 처리하지 않은 대조군의 Q4보다 3.6~4.5배 증가되어 엔도좀 라이소좀 성숙과정에 들어가고 있다고 볼 수 있다(Figure 8). 중요한 것은 G2보다 KKRK-G2, KKRKH-G2의 Q2 세포 비율이 각각 3.8배, 2.5배 증가되어 폴리머의 중량비가 2배 낮음에도 불구하고 엔도좀 탈출을 시도하는 비율이 높다는 것을 확인할 수 있다.
|
Figure 1 1H NMR data of (a) KKRK-G2; (b) KKRKH-G2. |
|
Figure 2 HPLC chromatograms of G2, KKRK-G2 and KKRKHG2. |
|
Figure 3 Complex test for polyplexes of (a) KKRK-G2; (b) KKRKH-G2 with pDNA. Control (pDNA only), the weight ratios of KKRK-G2 and KKRKH-G2 with pDNA. |
|
Figure 4 Cytotoxicity assay in HepG2 (a); NIH3T3 (b) using WST assay. The data of respective point indicates means±SD (n=4). |
|
Figure 5 Transfection efficiency of KKRK-G2 and KKRKH-G2 in (a) HepG2; (b) NIH3T3. The results are shown as mean±SD (n=3). |
|
Figure 6 Endocytosis inhibitor effects on transfection efficiency of the three polyplexes in NIH3T3 after 6 h. The results are shown as mean±SD (n=3). |
|
Figure 7 Internalization of KKRK-G2 polyplexes and KKRKH-G2 polyplexes in NIH3T3. Nucleus (Blue, Hoest 33342), pDNA (Red, Cyanine 3), Scale bar : 10 μm. |
|
Figure 8 AO assay for observation of endo-lysosomal disruption of NIH3T3 induced by G2, KKRK-G2, KKRKH-G2 using Flow cytometry. |
|
Table 1 Characterization of PAMAM Derivative/pDNA complexes |
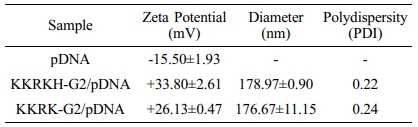
*Complex samples were prepared at 8:1 weight ratio and measured 3 times. |
SV40 바이러스, NOSTRINβ의 NLS 중 공통서열인 ‘KKRK’ 서열과 히스티딘을 이용하여 저분자량의 PAMAM 덴드리머에 결합하여 새로운 펩티드-고분자 하이브리드 고분자를 합성하였다. HepG2와 NIH3T3 세포주를 이용하여 공초점 형광현미경 및 루시페라아제 유전자 형질전환 실험을 통해서 KKRK-G2, KKRKH-G2는 대조군인 G2보다 pDNA 복합체들이 세포내부로의 전달이 매우 향상되었고, 또한 높은 형질변환 효율을 보여줌을 입증하였다. 합성물의 폴리플렉스는 약 170 nm의 나노 입자이며 충분한 양전하를 가져 높은 유전자 효율을 가졌지만, 고농도의 경우 세포 독성을 가지게 되었다. 세포 내 이입 저해 실험을 통해 거대음작용을 통한 메카니즘 가능성을 입증하였고, AO 실험을 통해 대조군에 비해 KKRK-G2, KKRKH-G2는 엔도좀 탈출 효율이 더 높다는 것을 확인하였다. 그러므로 KKRK-G2와 KKRKH-G2는 새로운 하이브리드 고분자로서 효율적인 비바이러스성 유전자 전달체로 활용할 수 있을 것이라 사료된다.
- 1. Global cell and gene therapy market: focus on products, applications, regions and competitive landscape – analysis and forecast, 2019-2025; BHP0729SA; BIS Research: California, July 2019.
- 2. Pillai, M.; Davies, M. M.; Thistlethwaite, F. C. Delivery of Adoptive Cell Therapy in the Context of the Health-care System in the UK: Challenges for Clinical Sites. Ther. Adv. Vaccines Immunother 2020, 8, 2515135520944355.
-

- 3. Supply Chain Optimization and Decentralized Manufacturing to Expand the Contract Cell and Gene Therapy Manufacturing Market, 2020-2026; MC16386737; Frost & Sullivan: California, October 2020.
- 4. Ramamoorth, M.; Narvekar, A. Non Viral Vectors in Gene Therapy- An Overview. J. Clin. Diagn. Res. 2015, 9, GE01-6.
-

- 5. Kaur, D.; Jain, K.; Mehra, N. K.; Kesharwani, P.; Jain, N. K. A Review on Comparative Study of PPI and PAMAM Dendrimers. J. Nanopart. Res. 2016, 18.
-

- 6. Shcharbin, D.; Janaszewska, A.; Klajnert-Maculewicz, B.; Ziemba, B.; Dzmitruk, V.; Halets, I.; Loznikova, S.; Shcharbina, N.; Milowska, K.; Ionov, M.; Shakhbazau, A.; Bryszewska, M. How to Study Dendrimers and Dendriplexes III. Biodistribution, Pharmacokinetics and Toxicity in vivo. J. Control. Release. 2014, 181, 40-52.
-

- 7. Boussif, O.; Lezoualch, F.; Zanta, M. A.; Mergny, M. D.; Scherman, D.; Demeneix, B.; Behr, J. P. A Versatile Vector for Gene and Oligonucleotide Transfer into Cells in Culture and in-Vivo - Polyethylenimine. Proc. Natl. Acad. Sci. USA 1995, 92, 7297-7301.
-

- 8. Jung, J.; Lee, J.; Kim, T. H.; Yang, B. S.; Lee, E.; Kim, Y. J.; Park, J. S.; Choi, J. S. PAMAM Dendrimer Conjugated with N-terminal Oligopeptides of Mouse Fibroblast Growth Factor 3 as a Novel Gene Carrier. Bull. Korean Chem. Soc. 2014, 35, 1036-1042.
-

- 9. Yu, G. S.; Yu, H. N.; Choe, Y. H.; Son, S. J.; Ha, T. H.; Choi, J. S. Sequential Conjugation of 6-Aminohexanoic Acids and L-Arginines to Poly(amidoamine) Dendrimer to Modify Hydrophobicity and Flexibility of the Polymeric Gene Carrier. Bull. Korean Chem. Soc. 2011, 32, 651-655.
-

- 10. Lee, J.; Kwon, Y. E.; Kim, Y.; Choi, J. S. Enhanced Transfection Efficiency of Low Generation PAMAM Dendrimer Conjugated with the Nuclear Localization Signal Peptide Derived from Herpesviridae. J. Biomat. Sci., Polym. Ed. 2021, 32, 22-41.
-

- 11. Lee, S.; Son, S. J.; Song, S. J.; Ha, T. H.; Choi, J. S. Polyamidoamine (PAMAM) Dendrimers Modified with Cathepsin-B Cleavable Oligopeptides for Enhanced Gene Delivery. Polymers 2017, 9.
-

- 12. Lee, J.; Lee, S.; Kwon, Y. E.; Kim, Y. J.; Choi, J. S. Gene Delivery by PAMAM Dendrimer Conjugated with the Nuclear Localization Signal Peptide Derived from Influenza B Virus Nucleoprotein. Macromol. Res. 2019, 27, 360-368.
-

- 13. Conti, E.; Uy, M.; Leighton, L.; Blobel, G.; Kuriyan, J. Crystallographic Analysis of the Recognition of a Nuclear Localization Signal by the Nuclear Import Factor Karyopherin Alpha. Cell 1998, 94, 193-204.
-

- 14. Leung, S. W.; Harreman, M. T.; Hodel, M. R.; Hodel, A. E.; Corbett, A. H. Dissection of the Karyopherin Alpha Nuclear Localization Signal (NLS)-binding Groove: Functional Requirements for NLS Binding. J. Biol. Chem. 2003, 278, 41947-53.
-

- 15. Kalderon, D.; Roberts, B. L.; Richardson, W. D.; Smith, A. E. A Short Amino Acid Sequence Able to Specify Nuclear Location. Cell 1984, 39, 499-509.
-

- 16. Wiesenthal, A.; Hoffmeister, M.; Siddique, M.; Kovacevic, I.; Oess, S.; Müller-Esterl, W.; Siehoff-Icking, A. NOSTRINbeta--a Shortened NOSTRIN Variant with a Role in Transcriptional Regulation. Traffic 2009, 10, 26-34.
-

- 17. Vaidyanathan, S.; Orr, B. G.; Holl, M. M. B. Role of Cell Membrane Vector Interactions in Successful Gene Delivery. Acc. Chem. Res. 2016, 49, 1486-1493.
-

- 18. Midoux, P.; Monsigny, M. Efficient Gene Transfer by Histidylated Polylysine pDNA Complexes. Bioconjugate Chem. 1999, 10, 406-411.
-

- 19. Lee, J.; Lee, S. H.; Choi, J. S., Enhanced Transfection Efficiency of PAMAM Dendrimer Conjugated with Amphiphilic Dipeptides in Gene Delivery. Polym. Korea 2018, 42, 813-821.
-

- 20. Shi, X. Y.; Bi, X. D.; Ganser, T. R.; Hong, S. P.; Myc, L. A.; Desai, A.; Holl, M. M. B.; Baker, J. R. HPLC Analysis of Functionalized Poly(amidoamine) Dendrimers and the Interaction Between a Folate-dendrimer Conjugate and Folate Binding Protein. Analyst 2006, 131, 842-848.
-

- 21. Chen, L. J.; Wang, H. B.; Li, X.; Nie, C.; Liang, T. B.; Xie, F. W.; Liu, K. J.; Peng, X. J.; Xie, J. P. Highly Hydrophilic Carbon Nanoparticles: Uptake Mechanism by Mammalian and Plant Cells. RSC Adv. 2018, 8, 35246-35256.
-

- 22. Yuan, Y. Y.; Zhang, C. J.; Liu, B. A Photoactivatable AIE Polymer for Light-Controlled Gene Delivery: Concurrent Endo/Lysosomal Escape and DNA Unpacking. Angew. Chem. Int. Ed. 2015, 54, 11419-11423.
-

- 23. Huhn, D.; Kantner, K.; Geidel, C.; Brandholt, S.; De Cock, I.; Soenen, S. J. H.; Gil, P. R.; Montenegro, J. M.; Braeckmans, K.; Mullen, K.; Nienhaus, G. U.; Klapper, M.; Parak, W. J. Polymer-Coated Nanoparticles Interacting with Proteins and Cells: Focusing on the Sign of the Net Charge. ACS Nano 2013, 7, 3253-3263.
-

- 24. Liu, Y. X.; Li, W.; Lao, F.; Liu, Y.; Wang, L. M.; Bai, R.; Zhao, Y. L.; Chen, C. Y. Intracellular Dynamics of Cationic and Anionic Polystyrene Nanoparticles without Direct Interaction with Mitotic Spindle and Chromosomes. Biomaterials 2011, 32, 8291-8303.
-

- 25. Sambale, F.; Wagner, S.; Stahl, F.; Khaydarov, R. R.; Scheper, T.; Bahnemann, D., Investigations of the Toxic Effect of Silver Nanoparticles on Mammalian Cell Lines. J. Nanomater. 2015, 2015.
-

- 26. Havrdova, M.; Hola, K.; Skopalik, J.; Tomankova, K.; Martin, P. A.; Cepe, K.; Polakova, K.; Tucek, J.; Bourlinos, A. B.; Zboril, R. Toxicity of Carbon Dots-Effect of Surface Functionalization on the Cell Viability, Reactive Oxygen Species Generation and Cell Cycle. Carbon 2016, 99, 238-248.
-

- 27. Moghimi, S. M.; Symonds, P.; Murray, J. C.; Hunter, A. C.; Debska, G.; Szewczyk, A. A Two-stage Poly(ethylenimine)-Mediated Cytotoxicity: Implications for Gene Transfer/therapy. Mol. Ther. 2005, 11, 990-995.
-

- 28. Lee, Y.; Lee, J.; Kim, M.; Kim, G.; Choi, J. S.; Lee, M. Brain Gene Delivery Using Histidine and Arginine-modified Dendrimers for Ischemic Stroke Therapy. J. Control. Release. 2021, 330, 907-919.
-

- 29. Barman, S.; Nayak, D. P. Lipid Raft Disruption by Cholesterol Depletion Enhances Influenza A Virus Budding from MDCK Cells. J. Virol. 2007, 81, 12169-78.
-

- 30. Kitchens, K. M.; Kolhatkar, I. B.; Swaan, P. W.; Ghandehari, H. Endocytosis Inhibitors Prevent Poly(amidoamine) Dendrimer Internalization and Permeability Across Ceco-2 Cells. Mol. Pharm. 2008, 5, 364-369.
-

- 31. Vidal, F.; Vasquez, P.; Diaz, C.; Nova, D.; Alderete, J.; Guzman, L. Mechanism of PAMAM Dendrimers Internalization in Hippocampal Neurons. Mol. Pharm. 2016, 13, 3395-3403.
-

- 32. Evangelatov, A.; Skrobanska, R.; Mladenov, N.; Petkova, M.; Yordanov, G.; Pankov, R. Epirubicin Loading in Poly(butyl cyanoacrylate) Nanoparticles Manifests via Altered Intracellular Localization and Cellular Response in Cervical Carcinoma (HeLa) Cells. Drug. Deliv. 2016, 23, 2235-2244.
-

- 33. Song, S. J.; Lee, S.; Lee, Y.; Choi, J. S. Enzyme-responsive Destabilization of Stabilized Plasmid-lipid Nanoparticles as an Efficient Gene Delivery. Eur. J. Pharm. Sci. 2016, 91, 20-30.
-

- Polymer(Korea) 폴리머
- Frequency : Bimonthly(odd)
ISSN 0379-153X(Print)
ISSN 2234-8077(Online)
Abbr. Polym. Korea - 2023 Impact Factor : 0.4
- Indexed in SCIE
 This Article
This Article
-
2022; 46(1): 113-121
Published online Jan 25, 2022
- 10.7317/pk.2022.46.1.113
- Received on Oct 14, 2021
- Revised on Oct 29, 2021
- Accepted on Nov 4, 2021
 Services
Services
Shared
 Correspondence to
Correspondence to
- Joon Sig Choi
-
Department of Biochemistry, College of Natural Sciences, Chungnam National University, 99 Daehak-ro, Daejeon 34134, Korea
- E-mail: joonsig@cnu.ac.kr
- ORCID:
0000-0002-8463-841X










 Copyright(c) The Polymer Society of Korea. All right reserved.
Copyright(c) The Polymer Society of Korea. All right reserved.